5.4 Cómputo en paralelo usando CPUs en un sistema de memoria compartida (SMC)
Contents
5.4 Cómputo en paralelo usando CPUs en un sistema de memoria compartida (SMC)#
Notas para contenedor de docker:
Comando de docker para ejecución de la nota de forma local:
nota: cambiar <ruta a mi directorio> por la ruta de directorio que se desea mapear a /datos dentro del contenedor de docker y <versión imagen de docker> por la versión más actualizada que se presenta en la documentación.
docker run --rm -v <ruta a mi directorio>:/datos --name jupyterlab_optimizacion_2 -p 8888:8888 -p 8787:8787 -d palmoreck/jupyterlab_optimizacion_2:<versión imagen de docker>
password para jupyterlab: qwerty
Detener el contenedor de docker:
docker stop jupyterlab_optimizacion_2
Documentación de la imagen de docker palmoreck/jupyterlab_optimizacion_2:<versión imagen de docker> en liga.
Nota generada a partir de liga1, liga2, liga3
Al final de esta nota la comunidad lectora:
Aprenderá a usar herramientas de software para cómputo en paralelo en C, Python y R.
Aprenderá características que tienen los sistemas de memoria compartida para la ejecución de códigos con cómputo en paralelo.
Se presentan códigos y sus ejecuciones en una máquina m4.16xlarge con una AMI ubuntu 20.04 - ami-042e8287309f5df03 de la nube de AWS. Se utilizó en la sección de User data el script_profiling_and_BLAS.sh
La máquina m4.16xlarge tiene las siguientes características:
%%bash
lscpu
Architecture: x86_64
CPU op-mode(s): 32-bit, 64-bit
Byte Order: Little Endian
Address sizes: 46 bits physical, 48 bits virtual
CPU(s): 64
On-line CPU(s) list: 0-63
Thread(s) per core: 2
Core(s) per socket: 16
Socket(s): 2
NUMA node(s): 2
Vendor ID: GenuineIntel
CPU family: 6
Model: 79
Model name: Intel(R) Xeon(R) CPU E5-2686 v4 @ 2.30GHz
Stepping: 1
CPU MHz: 2213.083
CPU max MHz: 3000.0000
CPU min MHz: 1200.0000
BogoMIPS: 4600.00
Hypervisor vendor: Xen
Virtualization type: full
L1d cache: 1 MiB
L1i cache: 1 MiB
L2 cache: 8 MiB
L3 cache: 90 MiB
NUMA node0 CPU(s): 0-15,32-47
NUMA node1 CPU(s): 16-31,48-63
Vulnerability Itlb multihit: KVM: Vulnerable
Vulnerability L1tf: Mitigation; PTE Inversion
Vulnerability Mds: Vulnerable: Clear CPU buffers attempted, no microcode; SMT Host state unknown
Vulnerability Meltdown: Mitigation; PTI
Vulnerability Spec store bypass: Vulnerable
Vulnerability Spectre v1: Mitigation; usercopy/swapgs barriers and __user pointer sanitization
Vulnerability Spectre v2: Mitigation; Full generic retpoline, STIBP disabled, RSB filling
Vulnerability Srbds: Not affected
Vulnerability Tsx async abort: Vulnerable: Clear CPU buffers attempted, no microcode; SMT Host state unknown
Flags: fpu vme de pse tsc msr pae mce cx8 apic sep mtrr pge mca cmov pat pse36 clflush mmx fxsr sse sse2 ht syscall nx pdpe1gb rdtscp lm constant_tsc arch_perfmon rep_good nopl xtopology nonstop_tsc cpuid aperfmperf pni pclmulqdq monitor est ssse3 fma cx16 pcid sse4_1 sse4_2 x2apic movbe popcnt tsc_deadline_timer aes xsave avx f16c rdrand hypervisor lahf_lm abm cpuid_fault invpcid_single pti fsgsbase bmi1 hle avx2 smep bmi2 erms invpcid rtm xsaveopt ida
%%bash
sudo lshw -C memory
*-firmware
description: BIOS
vendor: Xen
physical id: 0
version: 4.11.amazon
date: 08/24/2006
size: 96KiB
capabilities: pci edd
*-memory
description: System Memory
physical id: 1000
size: 256GiB
capabilities: ecc
configuration: errordetection=multi-bit-ecc
*-bank:0
description: DIMM RAM
physical id: 0
slot: DIMM 0
size: 16GiB
width: 64 bits
*-bank:1
description: DIMM RAM
physical id: 1
slot: DIMM 1
size: 16GiB
width: 64 bits
*-bank:2
description: DIMM RAM
physical id: 2
slot: DIMM 2
size: 16GiB
width: 64 bits
*-bank:3
description: DIMM RAM
physical id: 3
slot: DIMM 3
size: 16GiB
width: 64 bits
*-bank:4
description: DIMM RAM
physical id: 4
slot: DIMM 4
size: 16GiB
width: 64 bits
*-bank:5
description: DIMM RAM
physical id: 5
slot: DIMM 5
size: 16GiB
width: 64 bits
*-bank:6
description: DIMM RAM
physical id: 6
slot: DIMM 6
size: 16GiB
width: 64 bits
*-bank:7
description: DIMM RAM
physical id: 7
slot: DIMM 7
size: 16GiB
width: 64 bits
*-bank:8
description: DIMM RAM
physical id: 8
slot: DIMM 8
size: 16GiB
width: 64 bits
*-bank:9
description: DIMM RAM
physical id: 9
slot: DIMM 9
size: 16GiB
width: 64 bits
*-bank:10
description: DIMM RAM
physical id: a
slot: DIMM 10
size: 16GiB
width: 64 bits
*-bank:11
description: DIMM RAM
physical id: b
slot: DIMM 11
size: 16GiB
width: 64 bits
*-bank:12
description: DIMM RAM
physical id: c
slot: DIMM 12
size: 16GiB
width: 64 bits
*-bank:13
description: DIMM RAM
physical id: d
slot: DIMM 13
size: 16GiB
width: 64 bits
*-bank:14
description: DIMM RAM
physical id: e
slot: DIMM 14
size: 16GiB
width: 64 bits
*-bank:15
description: DIMM RAM
physical id: f
slot: DIMM 15
size: 16GiB
width: 64 bits
%%bash
uname -ar #r for kernel, a for all
Linux ip-10-0-0-84 5.4.0-1038-aws #40-Ubuntu SMP Fri Feb 5 23:50:40 UTC 2021 x86_64 x86_64 x86_64 GNU/Linux
Observación
En la celda anterior se utilizó el comando de magic %%bash. Algunos comandos de magic los podemos utilizar también con import. Ver ipython-magics
Sistemas de memoria compartida (SMC)#
Un SMC en general se ve como el siguiente dibujo:

El dibujo anterior es un SMC con acceso uniforme a la memoria (UMA), esto es, cada proceso o thread creado en el procesador o core accede con las mismas velocidades a la memoria.
La comunicación en este tipo de sistemas depende si se utilizan procesos o threads ya que aunque los procesos generados por un proceso principal tienen acceso a la memoria, los cambios/actualizaciones que haga uno de ellos a una variable no lo llegan a ver los otros procesos. Esto es distinto con los threads dado que un cambio que realice un thread en una variable sí lo pueden ver los otros threads. Lo anterior se debe a las distintas direcciones de memoria que utilizan los procesos vs la misma dirección de memoria que utilizan los threads.
Recuérdese que se crean threads a partir de un proceso principal, realizan fork, y una vez que terminan su ejecución se unen nuevamente, realizan join, esto es el threading.
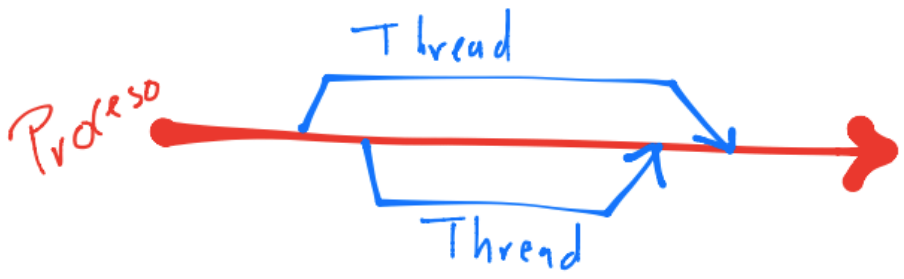
Ver Threading o Hyperthreading.
Observación
Hay paqueterías que crean subprocesos a partir de un proceso principal en lugar de threads. En este caso en lugar de un fork se realiza un spawn. Los subprocesos pueden ver cambios a variables hechos por otros.
Ver Contexts and start methods y difference between threadpool vs pool in python multiprocessing para diferencias entre fork y spawn.
Definición
Las variables que pueden ser accesadas por todos los threads en un SMC se les nombra variables compartidas.
Trabajar sobre SMC tiene ventajas y desventajas. Una ventaja es facilidad de comunicación y una desventaja es la coordinación para ejecutar instrucciones. Por ejemplo, con variables compartidas se puede realizar la comunicación entre los threads, sin embargo, para el uso de tales variables por diferentes threads debemos crear candados, locks. Lo anterior surge pues si un thread con etiqueta \(1\) accede a una variable compartida, otro thread con etiqueta \(2\), no podrá utilizarla hasta que el thread \(1\) finalice de usarla. Ver Thread Safety y Race Condition.
Definición
En una race condition múltiples threads intentan acceder a un recurso compartido, al menos uno de los accesos resulta en una modificación al recurso y posteriormente los siguientes accesos pueden no ver la modificación. A la sección del código que causa la race condition se le nombra critical section. Las critical sections se ejecutan con código secuencial o con locks para evitar las race conditions.
Comentarios
Para crear procesos o threads en los lenguajes de programación de C, Python o R, utilizamos las librerías, API’s o extensiones vía paquetes a tales lenguajes. Lo anterior se debe a que C, Python y R en sus implementaciones más utilizadas o estándar, fueron diseñados con el propósito de utilizarse sobre sistemas con un sólo procesador. En distintas implementaciones de los lenguajes hay soporte para SMC.
Ejemplos de máquinas con SMC son las laptops, los celulares, máquinas de escritorio con más de un core.
Otro tipo de SMC se puede representar con el siguiente dibujo:
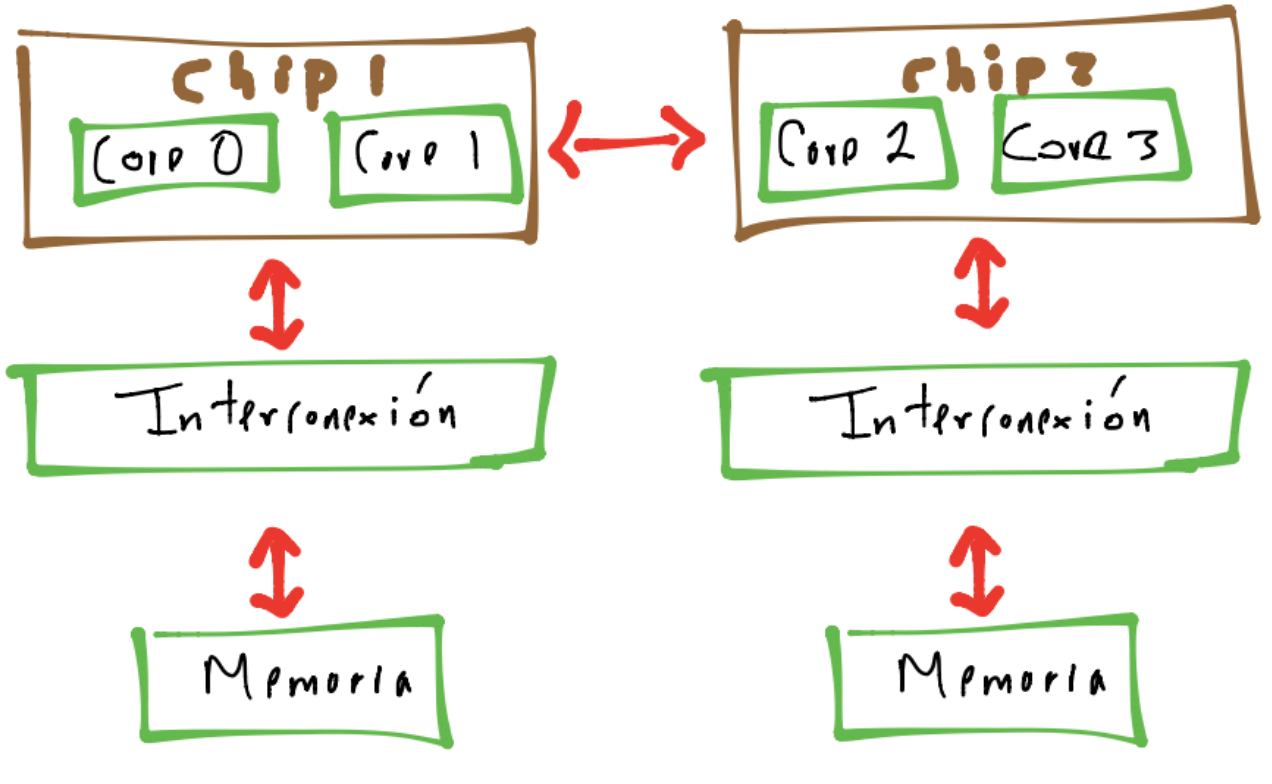
en el que los procesos o threads pueden acceder a la memoria en una forma no uniforme (NUMA). Una de las diferencias que se tienen entre un NUMA y un UMA es la tasa de transferencia de datos para cores que están más cercanos a una memoria.
Un sistema de memoria distribuida (SMD) tiene una conexión, por ejemplo vía una network, entre pares de core-memoria y en general se ve como el siguiente dibujo:
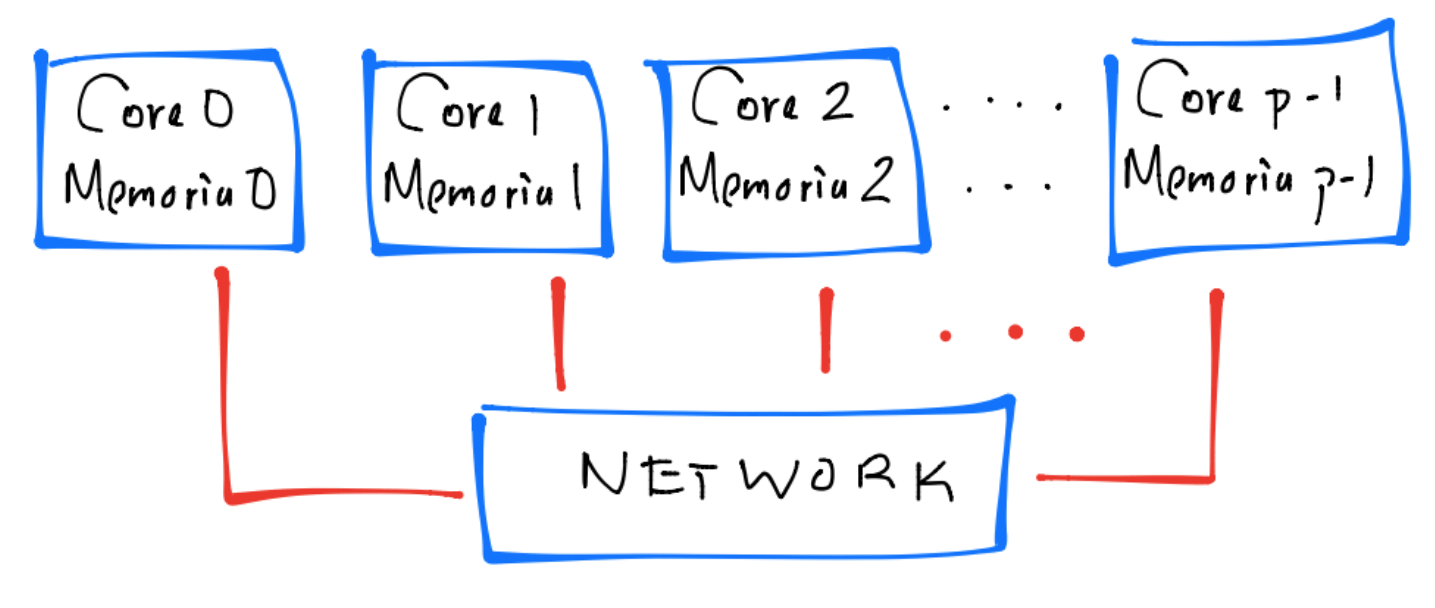
La memoria en un SMD asociada al core sólo puede ser accesada por éste y es inaccesible a los demás cores, esto es, se tiene una memoria “privada”. Lo anterior contrasta con los SMC en los que todos los cores accesan a una memoria compartida. En un SMD el cómputo distribuido crea múltiples procesos a partir de un proceso principal. En un SMC el cómputo paralelo crea múltiples procesos o threads. Un ejemplo de un SMD es un clúster de máquinas. Cada máquina en el clúster puede ser un SMC y por tanto los procesos en cada máquina tienen la capacidad de crear procesos o threads. En este caso se tiene un sistema híbrido SMD y SMC. Un programa diseñado para ejecutarse en un SMD puede ejecutarse en un SMC pues se divide su memoria de forma lógica en espacios de memoria privados para los threads.
OpenMP#
Es una extensión al lenguaje C y es una API para cómputo en paralelo en un SMC, aka, shared memory parallel programming con CPUs. Lo anterior OpenMP lo posibilita con el threading.
Las siglas MP se refieren a multiprocessing, un sinónimo de shared memory parallel computing. OpenMP se utiliza en un SMC por lo que cada thread tiene acceso a la memoria.
Algunas características de OpenMP son:
Paralelización de ciclos for secuenciales en los que las iteraciones son independientes una de la otra de forma simple.
Paralelización de tareas y sincronización explícita de threads.
Provee directivas vía
#pragma omppara el cómputo en paralelo en un SMC.
Comentario
Los pragma se utilizan para extender la funcionalidad de C pues no son parte de su especificación básica. Las versiones más recientes del compilador gcc soportan a los pragma y todas las preprocessor directives son por default de longitud una línea.
Ver pragmas.
Directiva parallel#
Ejemplo#
hello_world_omp.c
%%file hello_world_omp.c
#include<stdio.h>
#include<stdlib.h>
#include<omp.h>
void Hello(void);
int main(){
#pragma omp parallel
Hello();
return 0;
}
void Hello(void){
int my_rank = omp_get_thread_num();
int num_th = omp_get_num_threads();
printf("Hola del thread: %d de %d\n", my_rank, num_th);
}
Writing hello_world_omp.c
%%bash
gcc -Wall -fopenmp hello_world_omp.c -o hello_world_omp.out
%%bash
./hello_world_omp.out
Hola del thread: 60 de 64
Hola del thread: 9 de 64
Hola del thread: 6 de 64
Hola del thread: 13 de 64
Hola del thread: 44 de 64
Hola del thread: 15 de 64
Hola del thread: 12 de 64
Hola del thread: 16 de 64
Hola del thread: 18 de 64
Hola del thread: 14 de 64
Hola del thread: 62 de 64
Hola del thread: 17 de 64
Hola del thread: 45 de 64
Hola del thread: 48 de 64
Hola del thread: 23 de 64
Hola del thread: 55 de 64
Hola del thread: 24 de 64
Hola del thread: 42 de 64
Hola del thread: 26 de 64
Hola del thread: 49 de 64
Hola del thread: 0 de 64
Hola del thread: 29 de 64
Hola del thread: 50 de 64
Hola del thread: 34 de 64
Hola del thread: 53 de 64
Hola del thread: 47 de 64
Hola del thread: 19 de 64
Hola del thread: 39 de 64
Hola del thread: 38 de 64
Hola del thread: 36 de 64
Hola del thread: 33 de 64
Hola del thread: 37 de 64
Hola del thread: 43 de 64
Hola del thread: 59 de 64
Hola del thread: 28 de 64
Hola del thread: 21 de 64
Hola del thread: 46 de 64
Hola del thread: 27 de 64
Hola del thread: 25 de 64
Hola del thread: 31 de 64
Hola del thread: 32 de 64
Hola del thread: 41 de 64
Hola del thread: 20 de 64
Hola del thread: 22 de 64
Hola del thread: 35 de 64
Hola del thread: 58 de 64
Hola del thread: 3 de 64
Hola del thread: 4 de 64
Hola del thread: 2 de 64
Hola del thread: 1 de 64
Hola del thread: 5 de 64
Hola del thread: 7 de 64
Hola del thread: 61 de 64
Hola del thread: 10 de 64
Hola del thread: 8 de 64
Hola del thread: 11 de 64
Hola del thread: 51 de 64
Hola del thread: 40 de 64
Hola del thread: 63 de 64
Hola del thread: 30 de 64
Hola del thread: 57 de 64
Hola del thread: 52 de 64
Hola del thread: 54 de 64
Hola del thread: 56 de 64
Comentarios
omp.hes un header file con prototipos y definiciones de macros para uso de la librería de funciones y macros de OpenMP.La función
Helloserá ejecutada por los threads.La función
omp_get_thread_numda el rank asignado por el run time system a cada thread.Con la función
omp_get_num_threadsse obtiene el número de threads que realizaron un fork del thread principal.Obsérvese que para la compilación se utilizó la flag
-fopenmppara soporte de OpenMP.Dependiendo del número de cores de nuestro sistema tendremos diferentes número de
printf’s.Lo que continúa a la línea de
#pragma omp paralleles un structured block, esto es, un statement o conjunto de statements que tienen un punto de entrada y un punto de salida. En el caso anterior sólo se llama a la funciónHello.No se permiten statements como el siguiente:
#pragma omp parallel
if(...) break;
ni tampoco:
#pragma omp parallel
{
if(variable == valor) return 1;
return -1;
}
A continuación de la directiva parallel podemos usar diferentes tipos de clauses. Una clause en OpenMP es un texto que modifica una directiva. Por ejemplo, podemos usar la clause num_threads para especificar el número de threads que ejecutarán el structured block.
num_threads clause#
Ejemplo#
hello_world_omp_num_threads.c
%%file hello_world_omp_num_threads.c
#include<stdio.h>
#include<stdlib.h>
#include<omp.h>
void Hello(void);
int main(){
int n_threads = 5;
#pragma omp parallel num_threads(n_threads)
Hello();
return 0;
}
void Hello(void){
int my_rank = omp_get_thread_num();
int num_th = omp_get_num_threads();
printf("Hola del thread: %d de %d\n", my_rank, num_th);
}
Writing hello_world_omp_num_threads.c
%%bash
gcc -Wall -fopenmp hello_world_omp_num_threads.c -o hello_world_omp_num_threads.out
%%bash
./hello_world_omp_num_threads.out
Hola del thread: 0 de 5
Hola del thread: 4 de 5
Hola del thread: 1 de 5
Hola del thread: 3 de 5
Hola del thread: 2 de 5
Reduction clause#
OpenMP provee la reduction clause para aplicar la operación de suma (operador binario) a cada resultado calculado de un thread de forma repetida y almacenar en una variable la respuesta.
Nombramos reduction variable a la variable que almacenará los resultados intermedios calculados por cada thread y reduction operator a la operación binaria (por ejemplo una suma o multiplicación) que se aplica repetidamente a una secuencia de operandos para obtener un resultado, en un proceso que se le nombra reduction.
Por ejemplo, si A es un arreglo de n enteros, el cálculo:
int sum = 0;
for(i=0;i<n;i++)
sum += A[i];
es un proceso de reduction en el que el reduction operator es la suma.
En openMP utilizamos la reduction clause en la parallel directive.
sum_shared = 0.0
#pragma omp parallel num_threads(n_threads) reduction(+: sum_shared)
sum_shared += Rcf_parallel(a,h_hat,n_subintervals_per_thread);
Con la reduction clause openMP crea una variable privada por cada thread y el run time system almacena el resultado de cada thread en esta variable. openMP también crea una critical section y los valores almacenados en las variables privadas son sumadas en esta critical section y almacenados en la reduction variable sum_shared. El reduction operator es: +.
La sintaxis de la reduction clause es:
reduction( <operator>: <variable list>)
El reduction operator puede ser cualquiera de los operadores: +,*,-,&,|,^,&&,||. Cabe señalar que el proceso de reduction asume que los operadores utilizados cumplen con la propiedad asociativa (por ejemplo, el operador de resta no cumple con esto).
La variable que está en la reduction clause es compartida. Sin embargo una variable privada es creada por cada thread en el team (con el mismo nombre que aparece en la reduction clause) y si un thread ejecuta un statement en el parallel block que involucra a la variable, entonces se utiliza la variable privada. Al finalizar el parallel block, los valores calculados en las variables privadas son combinados en la variable compartida.
Ejemplo#
En el siguiente código se muestra el uso de nombres de variables al definir variables privadas y compartidas en una reduction clause.
private_shared_variable_reduction_clause_example.c
%%file private_shared_variable_reduction_clause_example.c
#include<stdio.h>
#include<stdlib.h>
#include<omp.h>
int main(int argc, char *argv[]){
long n_threads;
int private_variable;
int sum_shared = 7;
n_threads = strtol(argv[1], NULL, 10);
printf("variable sum_shared al inicio : %d\n", sum_shared);
#pragma omp parallel num_threads(n_threads) reduction(+: sum_shared)
{
int my_rank = omp_get_thread_num();
if(my_rank==0)
printf("sum_shared, printf 1 del thread 0: %d\n", sum_shared);
else
printf("sum_shared, prinftf 1 thread 1: %d\n", sum_shared);
sum_shared = (my_rank==0)?10:20;
if(my_rank==0)
printf("sum_shared, printf 2 del thread 0: %d\n", sum_shared);
else
printf("sum_shared, printf 2 del thread 1: %d\n", sum_shared);
private_variable = (my_rank==0)?1:2;
sum_shared += private_variable;
if(my_rank==0)
printf("sum_shared, printf 3 del thread 0: %d\n", sum_shared);
else
printf("sum_shared, printf 3 del thread 1: %d\n", sum_shared);
}
printf("sum_shared al final de la directive parallel %d\n", sum_shared);
return 0;
}
Writing private_shared_variable_reduction_clause_example.c
Compilamos:
%%bash
gcc -Wall -fopenmp private_shared_variable_reduction_clause_example.c -o private_shared_variable_reduction_clause_example.out
Ejecutamos:
%%bash
./private_shared_variable_reduction_clause_example.out 2
variable sum_shared al inicio : 7
sum_shared, printf 1 del thread 0: 0
sum_shared, printf 2 del thread 0: 10
sum_shared, printf 3 del thread 0: 11
sum_shared, prinftf 1 thread 1: 0
sum_shared, printf 2 del thread 1: 20
sum_shared, printf 3 del thread 1: 22
sum_shared al final de la directive parallel 40
Comentario
La variable sum_shared se inicializa en 0. En general, las variables privadas creadas para una reduction clause son inicializadas al identity value para el operator. Por ejemplo, si el operator es la multiplicación, entonces las variables privadas son inicializadas en 1.
Ejemplo regla compuesta del rectángulo#
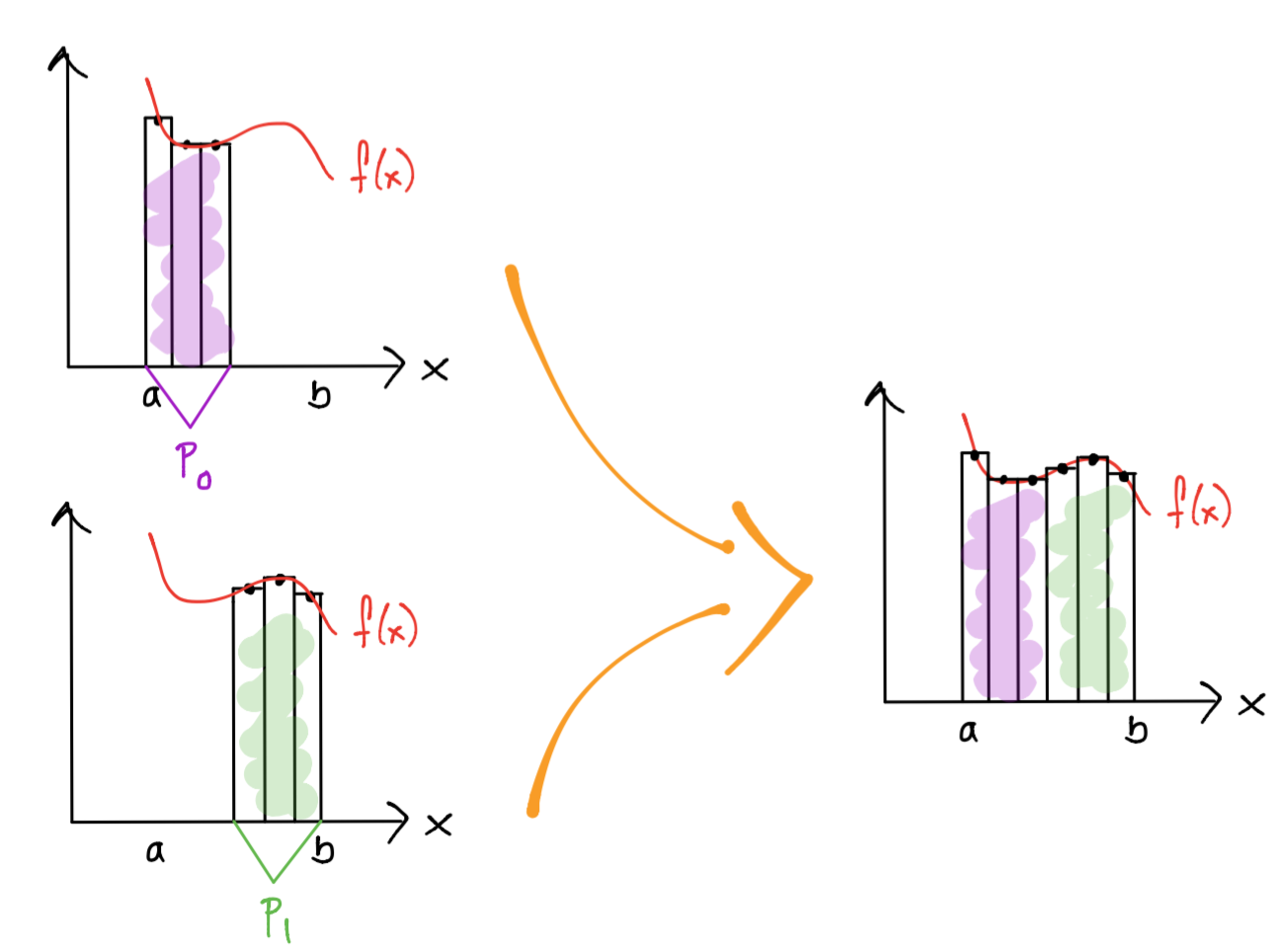
1.Partitioning: la tarea a realizar es el cálculo de un área de un rectángulo para un subintervalo.
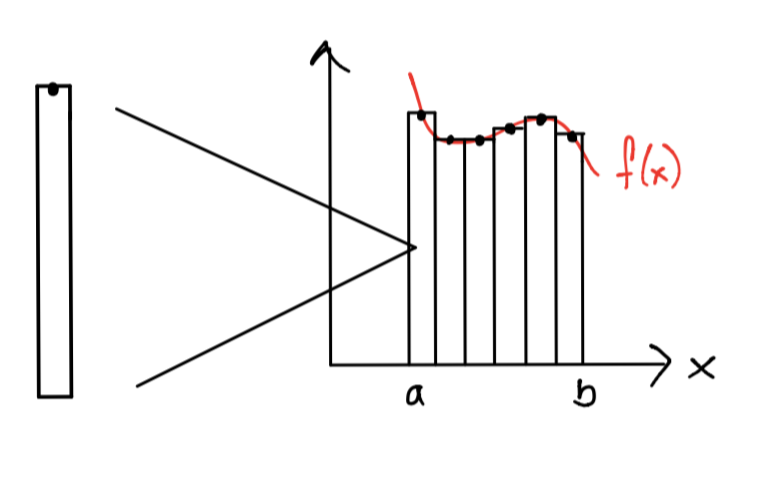
2.Communication y mapping: los subintervalos deben repartirse entre los cores y se debe comunicar esta repartición por algún medio (por ejemplo con variables en memoria).
3.Aggregation: un core puede calcular más de un área de un rectángulo si recibe más de un subintervalo.
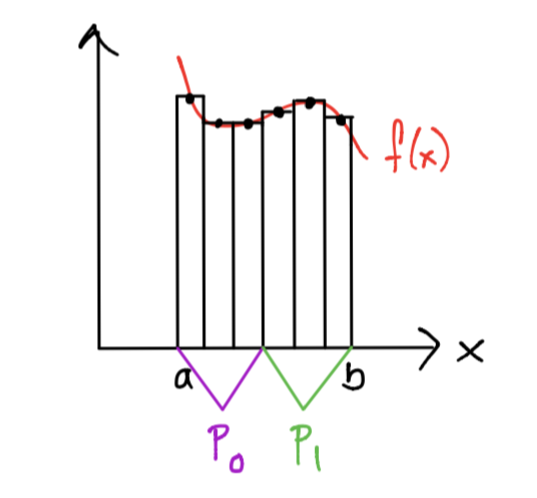
4.Communication y mapping: el área de los rectángulos calculados por cada procesador deben sumarse para calcular la aproximación a la integral.
Para la medición de tiempos se utilizaron las ligas: measuring-time-in-millisecond-precision y find-execution-time-c-program.
Rcf_parallel.c
%%file Rcf_parallel.c
#include<stdio.h>
#include<stdlib.h>
#include<omp.h>
#include<math.h>
#include<time.h>
#include <sys/time.h>
double Rcf(double a, double h_hat, int n_s_t);
double f(double node);
int main(){
double sum_shared = 0.0; //shared variable for threads
double a = 0.0, b = 1.0;
int n = 1e7; //number of subintervals
double h_hat = (b-a)/n;
int n_subintervals_per_thread; //number of subintervals assigned to each thread
int n_threads[7] = {0};//each entry of n_threads must divide n variable exactly
int len_n_threads_array = 0;
double obj = 0.7468241328124271;
struct timeval start;
struct timeval end;
long seconds;
long long mili;
int i;
n_threads[0] = 1;
n_threads[1] = 2;
n_threads[2] = 4;
n_threads[3] = 8;
n_threads[4] = 16;
n_threads[5] = 32;
n_threads[6] = 64;
len_n_threads_array = sizeof(n_threads)/sizeof(n_threads[0]);
for(i=0;i<len_n_threads_array;i++){
n_subintervals_per_thread = n/n_threads[i];
gettimeofday(&start, NULL);
#pragma omp parallel num_threads(n_threads[i]) reduction(+: sum_shared)
sum_shared += Rcf(a,h_hat,n_subintervals_per_thread);
sum_shared = h_hat*sum_shared;
gettimeofday(&end, NULL);
seconds = (end.tv_sec - start.tv_sec);
mili = 1000*(seconds) + (end.tv_usec - start.tv_usec)/1000;
printf("Integral de %f a %f = %1.15e\n", a,b,sum_shared);
printf("Error relativo de la solución: %1.15e\n", fabs(sum_shared-obj)/fabs(obj));
printf("Tiempo de ejecución con %d threads: %lld miliseconds\n", n_threads[i],mili);
printf("----------------------\n");
sum_shared = 0.0;
}
return 0;
}
double Rcf(double a, double h_hat, int n_s_t){
int begin, end;
int my_rank = omp_get_thread_num();
double local_int=0;
int i;
double x;
begin = my_rank*n_s_t;
end = begin + n_s_t;
for(i=begin;i<=end-1;i++){
x = a+(i+1/2.0)*h_hat;
local_int += f(x);
}
return local_int;
}
double f(double node){
double f_value;
f_value = exp(-pow(node,2));
return f_value;
}
Writing Rcf_parallel.c
%%bash
gcc -Wall -fopenmp Rcf_parallel.c -o Rcf_parallel.out -lm
%%bash
./Rcf_parallel.out
Integral de 0.000000 a 1.000000 = 7.468241328123898e-01
Error relativo de la solución: 4.994950214975770e-14
Tiempo de ejecución con 1 threads: 492 miliseconds
----------------------
Integral de 0.000000 a 1.000000 = 7.468241328124163e-01
Error relativo de la solución: 1.441994556109076e-14
Tiempo de ejecución con 2 threads: 239 miliseconds
----------------------
Integral de 0.000000 a 1.000000 = 7.468241328124452e-01
Error relativo de la solución: 2.423145491193603e-14
Tiempo de ejecución con 4 threads: 121 miliseconds
----------------------
Integral de 0.000000 a 1.000000 = 7.468241328124374e-01
Error relativo de la solución: 1.382530863073651e-14
Tiempo de ejecución con 8 threads: 60 miliseconds
----------------------
Integral de 0.000000 a 1.000000 = 7.468241328124300e-01
Error relativo de la solución: 3.865140047302680e-15
Tiempo de ejecución con 16 threads: 30 miliseconds
----------------------
Integral de 0.000000 a 1.000000 = 7.468241328124294e-01
Error relativo de la solución: 3.121843884359856e-15
Tiempo de ejecución con 32 threads: 21 miliseconds
----------------------
Integral de 0.000000 a 1.000000 = 7.468241328124262e-01
Error relativo de la solución: 1.189273860708517e-15
Tiempo de ejecución con 64 threads: 26 miliseconds
----------------------
Comentarios
Dividimos el número de subintervalos
nentre el número de threads que deseamos lanzar, por esto,ndebe ser divisible entre las entradas del arrayn_threads. Esta cantidad es el número de subintervalos contiguos que le corresponden a cada thread.Además, se debe de agregar el resultado de la suma de cada thread. Esto es posible realizar de forma sencilla definiendo una variable que sea compartida. Al definir tal variable en la función main y antes de un parallel block el default es que sea considerada como compartida.
Las variables que son privadas se definen en la función
Rcf.
Directiva parallel for#
Esta directiva ayuda a paralelizar ciclos for que tienen una forma canónica como sigue:

La directiva realiza un fork de threads en un team para ejecutar el structured block que continúa a la directiva. Tal structured block debe ser un ciclo for.
Con esta directiva se dividen las iteraciones del loop entre los threads.
Por default todas las variables definidas antes de la directiva parallel son compartidas pero en la directiva parallel for, el índice del ciclo for es privada, cada thread tiene su propia copia de tal índice.
Ejemplo regla compuesta del rectángulo con directiva parallel for#
%%file Rcf_parallel_for.c
#include<stdio.h>
#include<stdlib.h>
#include<math.h>
#include<time.h>
#include <sys/time.h>
double Rcf(long n_t, double ext_izq, double ext_der, int n,\
double s_res);
double f(double nodo);
int main(int argc, char *argv[]){
double sum_res = 0.0;
double a = 0.0, b = 1.0;
int n = 1e7;
struct timeval start;
struct timeval end;
long seconds;
long long mili;
long n_threads;
double res_rcf;
double obj = 0.7468241328124271;
n_threads = strtol(argv[1], NULL, 10);
gettimeofday(&start, NULL);
res_rcf = Rcf(n_threads, a,b,n, sum_res);
gettimeofday(&end, NULL);
seconds = (end.tv_sec - start.tv_sec);
mili = 1000*(seconds) + (end.tv_usec - start.tv_usec)/1000;
printf("Integral de %f a %f = %1.15e\n", a,b,res_rcf);
printf("Error relativo de la solución: %1.15e\n", fabs(res_rcf-obj)/fabs(obj));
printf("Tiempo de ejecución: %lld milisegundos", mili);
return 0;
}
double Rcf(long n_th, double a, double b, int n, double sum_res){
double h_hat = (b-a)/n;
double x = 0.0;
int i = 0;
sum_res = 0.0;
# pragma omp parallel for num_threads(n_th) reduction(+: sum_res)
for(i = 0; i <= n-1; i++){
x = a+(i+1/2.0)*h_hat;
sum_res += f(x);
}
return h_hat*sum_res;
}
double f(double nodo){
double valor_f;
valor_f = exp(-pow(nodo,2));
return valor_f;
}
Writing Rcf_parallel_for.c
%%bash
gcc -Wall -fopenmp Rcf_parallel_for.c -o Rcf_parallel_for.out -lm
%%bash
./Rcf_parallel_for.out 1
Integral de 0.000000 a 1.000000 = 7.468241328123898e-01
Error relativo de la solución: 4.994950214975770e-14
Tiempo de ejecución: 489 milisegundos
%%bash
./Rcf_parallel_for.out 2
Integral de 0.000000 a 1.000000 = 7.468209392885364e-01
Error relativo de la solución: 4.276139120784953e-06
Tiempo de ejecución: 268 milisegundos
%%bash
./Rcf_parallel_for.out 4
Integral de 0.000000 a 1.000000 = 7.468245048387456e-01
Error relativo de la solución: 4.981444789795125e-07
Tiempo de ejecución: 196 milisegundos
%%bash
./Rcf_parallel_for.out 8
Integral de 0.000000 a 1.000000 = 7.468233188076527e-01
Error relativo de la solución: 1.089955102679663e-06
Tiempo de ejecución: 172 milisegundos
%%bash
./Rcf_parallel_for.out 16
Integral de 0.000000 a 1.000000 = 7.468247754499342e-01
Error relativo de la solución: 8.604937613327165e-07
Tiempo de ejecución: 190 milisegundos
%%bash
./Rcf_parallel_for.out 32
Integral de 0.000000 a 1.000000 = 7.468178324971565e-01
Error relativo de la solución: 8.436143120972810e-06
Tiempo de ejecución: 427 milisegundos
Ejercicio
Implementar la regla de Simpson compuesta con OpenMP utilizando una reduction clause y la directiva parallel for en una máquina de AWS con las mismas características que la que se presenta en esta nota y medir tiempo de ejecución.
Multiprocessing#
El módulo Multiprocessing permite realizar procesamientos basados en procesos o threads para compartir trabajo, datos y lidiar con que la implementación estándar de Python, CPython no utiliza múltiples cores por default para procesamiento.
En el apartado de Cpython-Design se menciona el por qué CPython no soporta ejecución multithreaded o multiprocesses y a continuación se colocan unos párrafos de tal discusión.
A particular feature of CPython is that it makes use of a global interpreter lock (GIL) on each CPython interpreter process, which means that within a single process, only one thread may be processing Python bytecode at any one time. This does not mean that there is no point in multithreading; the most common multithreading scenario is where threads are mostly waiting on external processes to complete.
For example, imagine when three threads are servicing separate clients. One thread may be waiting for a client to reply, and another may be waiting for a database query to execute, while the third thread is actually processing Python code.
However, the GIL does mean that CPython is not suitable for processes that implement CPU-intensive algorithms in Python code that could potentially be distributed across multiple cores. …
Se recomienda usar este módulo para el shared memory programming y para trabajos que son demandantes de CPU. No se recomienda su uso en trabajos demandantes en I/O. En su lugar se tienen paquetes de Python para ejecución del código de forma asíncrona como asyncio o tornado. También Dask permite tal tipo de ejecución, ver dask-asynchronous.
Comentario
Otro módulo en Python para procesamiento en un SMC con CPUs es concurrent.futures que provee el comportamiento principal de Multiprocessing. Ver concurrent-futures-processpoolexecutor-vs-multiprocessing-pool-pool y concurrent-futures-vs-multiprocessing-in-python-3 para más sobre concurrent.futures y concurrent.futures vs multiprocessing.
Nota sobre el GIL y multiprocessing#
Aunque en Python los threads son nativos del sistema operativo (esto es, no se simulan, son realmente threads del sistema operativo creados en el hardware), están limitados por el global interpreter lock (GIL), de modo que un sólo thread interactúe con un objeto Python en un único tiempo. Esto degrada el performance de los programas pues si un thread con etiqueta 1 tiene el GIL entonces otro thread con etiqueta 2 esperará hasta que el thread 1 lo suelte. Ver Understanding the Python GIL.
Al usar el módulo Multiprocessing ejecutamos en paralelo un número de intérpretes Python (CPython), cada uno con su propio espacio de memoria privada y su propio GIL.
Observación
En multiprocessing se utilizan subprocesos en lugar de threads y en lugar de fork se realiza spawn.
Ejemplo#
En Multiprocessing los procesos son generados al utilizar la clase Process para crear objetos y llamar al método start. Ver Process para documentación de ésta clase.
from multiprocessing import Process
def f():
print('hello world! de subproceso')
p1 = Process(target=f)
p2 = Process(target=f)
p1.start()
p2.start()
p1.join()
p2.join()
print('hello world! de proceso')
hello world! de subproceso
hello world! de subproceso
hello world! de proceso
Comentarios
startsólo puede ser llamada una vez por objetoProcess.Con el primer
joinel proceso principal espera a que terminep1. Con el segundojoinel proceso principal espera a que terminep2.
Observación
Es buena práctica explícitamente hacer joins para cada objeto process que realizó start. Ver Programming guidelines para buenas prácticas.
La clase Process recibe la función a ejecutar para cada proceso con el argumento target y también tiene args para los argumentos de la función:
def f(s):
print(s)
p1 = Process(target=f, args=('hola',))
p2 = Process(target=f, args=('mundo',))
p1.start()
p2.start()
p1.join()
p2.join()
hola
mundo
Comentarios
De acuerdo a Programming guidelines se debe usar un bloque del tipo
if __name__=='__main__':para las personas que tienen Windows puedan ejecutar sin problema el código:
if __name__=='__main__':
p1 = Process(target=f, args=('hola',))
p2 = Process(target=f, args=('mundo',))
p1.start()
p2.start()
p1.join()
p2.join()
El bloque del if ayuda a que los subprocesos importen el módulo __main__ (por lo que no se ejecuta la sección que está dentro de if __name__=='__main__': pues no son programas principales) y continúa la ejecución de las líneas de start (cada subproceso ejecuta f) y join. Sin tal bloque if, el notebook quedará bloqueado pues una celda con el código anterior creará subprocesos que a su vez crearán otros subprocesos, que a su vez crearán otros subprocesos… y así de forma recursiva.
Los argumentos en
argsdeProcesstienen que ser objetos pickable o serializados. Ver what can be pickled and unpickled para una lista de objetos pickable.
En Multiprocessing tenemos la función cpu_count para determinar el número de cores que el sistema operativo puede usar. Este número es la cantidad física o simulada (hyperthreading) de cores.
import multiprocessing
print(multiprocessing.cpu_count())
64
Pool of workers#
La clase Pool crea un conjunto (pool) de procesos tipo worker que procesarán las tareas a realizar vía funciones tipo map o apply. Se hace map del input hacia los procesadores y se colecta el output de éstos. Mientras el map se realiza, el proceso que lanzó el map se bloquea hasta que finalicen las tareas (aunque hay map_async). El output es una lista.
Observación
El procesamiento de las tareas podríamos hacerlo con la clase Process de arriba pero tendríamos que utilizar un ciclo y colectar los resultados.
Comentario
Para un gran número de tareas a realizar, utilizar Pool. Para pocas tareas (menos de \(10\)) a realizar, utilizar Process.
Ejemplo#
from multiprocessing import Pool
def f(dummy):
return 'hello world!'
pool = Pool(multiprocessing.cpu_count())
results = pool.map(f,range(multiprocessing.cpu_count()))
print(results)
pool.close()
pool.join()
['hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!', 'hello world!']
def f(dummy):
return 'hello world!'
num_processes=2
pool = Pool(num_processes)
results = pool.map(f,range(num_processes))
print(results)
pool.close()
pool.join()
['hello world!', 'hello world!']
Ejemplo utilizando apply#
def f():
return 'hello world!'
num_processes=2
pool = Pool(num_processes)
results = [pool.apply(f) for x in range(num_processes)]
print(results)
pool.close()
pool.join()
['hello world!', 'hello world!']
Ejemplo con context manager#
Nos ayuda a no escribir statements: pool.close, pool.join.
def f(dummy):
return 'hello world!'
num_processes=2
with Pool(processes=num_processes) as pool:
results = pool.map(f,range(num_processes))
print(results)
['hello world!', 'hello world!']
Ejemplo para pasar múltiples argumentos vía starmap#
def f(s1, s2):
return s1 + s2
num_processes=2
with Pool(processes=num_processes) as pool:
iterable = [("hola"," mundo 1arg"),("hola", " mundo 2arg")]
results = pool.starmap(f, iterable)
print(results)
['hola mundo 1arg', 'hola mundo 2arg']
Ejemplo regla compuesta del rectángulo#
import math
import time
from scipy.integrate import quad
from pytest import approx
f = lambda x: math.exp(-x**2)
a = 0
b = 1
n = 10**7
h_hat = (b-a)/n
p = multiprocessing.cpu_count()
n_subintervals_per_subprocess = int(n/p)
Comentario
n_subintervals_per_subprocess es el número de subintervalos por core. Se asume que n es divisible por p. Si no se cumple esto, se puede definir n_subintervals_per_subprocess = int(n/p) habiendo definido n primero y redefinir n como: n = p*n_subintervals_per_subprocess
print("número de subintervalos:", n)
número de subintervalos: 10000000
print("número de subintervalos por proceso:", n_subintervals_per_subprocess)
número de subintervalos por proceso: 156250
def Rcf_multiprocessing(my_id):
begin = my_id*n_subintervals_per_subprocess
end = begin+n_subintervals_per_subprocess
h_hat = (b-a)/n
sum_res = 0
for i in range(begin,end):
x = a+(i+1/2)*h_hat
sum_res += f(x)
return sum_res
pool = Pool(processes=p)
start_time=time.time()
res_map = pool.map(Rcf_multiprocessing,range(p))
res_multiprocessing = h_hat*sum(res_map)
end_time = time.time()
secs = end_time-start_time
print("Rcf_multiprocessing tomó",secs,"segundos" )
Rcf_multiprocessing tomó 0.16410470008850098 segundos
obj, err = quad(f, a, b)
print(res_multiprocessing == approx(obj))
True
%%timeit -n 5 -r 10 -o
res_map = pool.map(Rcf_multiprocessing,range(p))
res_multiprocessing = h_hat*sum(res_map)
135 ms ± 6.22 ms per loop (mean ± std. dev. of 10 runs, 5 loops each)
<TimeitResult : 135 ms ± 6.22 ms per loop (mean ± std. dev. of 10 runs, 5 loops each)>
Rcf_multiprocessing_timeit = _
print(Rcf_multiprocessing_timeit.average)
0.1347037914800006
pool.close()
pool.join()
Dask#
Permite procesamiento en SMC o SMD.
Extiende interfaces de arrays, dataframes y listas usadas en NumPy, Pandas e iterators para su procesamiento. En el contexto de Dask a los tipos de valores anteriores les denomina big data collections.
Manejo de datasets que no caben en la memoria RAM: larger than memory datasets o out of core datasets.
Las big data collections soportadas por Dask son alternativas a los arrays y dataframes de NumPy y Pandas respectivamente para datasets grandes.
Provee ejecución con cómputo paralelo o distribuido de algunas funciones de NumPy y Pandas.
Soporta un task scheduling dinámico y optimizado para cómputo.
Comentarios
Task scheduling es un enfoque de paralelización en el que dividimos el programa en muchos tasks medium-sized. En Dask se representan tales tasks como nodos de un grafo con líneas entre éstos si un task depende de lo producido por otro.
Dinámico pues las task graphs pueden ser definidas a partir de las big data collections o por users.
Dask posee módulos para optimizar la ejecución de las task graphs (más abajo se describe cómo construir una task graph). Ver optimization.
En Dask existen task schedulers para ejecución en paralelo, distribuida o secuencial de la task graph.
Soporta el enfoque de paralelización de datos con las collections:
¿Cómo se implementan una task y una task/dask graph en Dask?#
Cada task se implementa como una tupla de Python que contiene funciones y argumentos de las mismas.
def fun_sum(arg1,arg2):
return arg1+arg2
t = (fun_sum, -1, 2)
print(t)
(<function fun_sum at 0x7fd2dfffa430>, -1, 2)
Una task graph o dask graph se implementa como un diccionario de tasks:
dic = {"arg1": -1,
"arg2": 2,
"res": (fun_sum, "arg1", "arg2")}
¿Cómo ejecutamos la task/dask graph?#
Con los schedulers definidos en Dask. Ver Scheduling y Scheduler Overview
import dask
Para cualquier scheduler definido en dask el entry point es una función get la cual recibe una task/dask graph y una key o lista de keys para cálculos:
print(dask.get(dic, "res")) #synchronous scheduler
1
print(dask.threaded.get(dic, "res")) #scheduler backed by a thread pool
1
print(dask.multiprocessing.get(dic, "res")) #scheduler backed by a process pool
1
Comentarios
La función
dask.getejecuta con unsynchronous schedulerel cual sólo utiliza un thread de ejecución sin paralelización. Útil para debugging y perfilamiento.La función
dask.threaded.getejecuta con un schedulermultiprocessing.pool.ThreadPool. Se hacen forks y joins de un proceso. El overhead para ejecución es pequeño y no hay costo en transferencia de datos entre los tasks. Sin embargo debido al GIL de Python, este scheduler provee paralelización si tu código es esencialmente “no Python”, por ejemplo si utilizas código de NumPy, Pandas o Cython.La función
dask.multiprocessing.getejecuta con un schedulermultiprocessing.Pool. Para cada task se crea un proceso, no tiene problemas del GIL de Python para código Python. Sin embargo, mover datos hacia procesos y de vuelta al proceso principal tiene costos altos. Útil para tasks que no requieren mucha transferencia de datos y los inputs y outputs son pequeños en tamaño.En la documentación de Dask se recomienda utilizar al scheduler distributed en lugar de usar
dask.multiprocesing.get. La documentación se encuentra en Dask.distributed. Para una pequeña explicación ver Dask Distributed local.
¿Cómo visualizamos la task/dask graph?#
Usamos visualize. Ver visualize task graphs y dask.visualize.
dask.visualize(dic,"res")
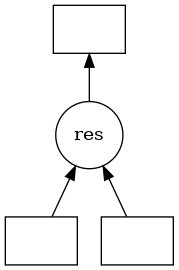
Las cajas representan datos, los círculos representan funciones que se ejecutan sobre tales datos y las flechas especifican cuáles funciones producen/consumen qué datos.
Flujo de trabajo con Dask collections#
En resumen en Dask seguimos un flujo como el siguiente dibujo.
Observación
Para crear task/dask graphs podemos usar las collections o bien, definir nuestras propias.
Comentario
Si trabajamos con las collections será extraño que trabajemos a nivel de funciones get. Cada collection tiene un default scheduler y una función compute que calcula el output de la collection.
Ejemplo#
dask arrays y dask dataframes utilizan al threaded scheduler por default pero con compute puede cambiarse de scheduler.
import dask.array as da
x = da.arange(100)
print(x)
dask.array<arange, shape=(100,), dtype=int64, chunksize=(100,), chunktype=numpy.ndarray>
x
|
print(type(x))
<class 'dask.array.core.Array'>
Llamamos a la función dask.compute.
print(x.sum().compute())
4950
Podemos cambiar de scheduler especificando en compute:
print(x.sum().compute(scheduler="processes"))
4950
Por default dask array trabaja con chunks.
x = da.arange(100, chunks=10)
x
|
Por default, dask array trabaja de una forma lazy, esto es, dask array no calculará ningún resultado hasta que explícitamente se le dé la instrucción.
y = x.sum()
y
|
Hasta este punto no se ha calculado la suma (cuyo resultado es un escalar). Después de llamar a compute se ejecuta la task graph para ejecución en paralelo (si se definió al scheduler para este tipo de ejecución).
print(y.compute())
4950
Y podemos visualizar la task/dask graph definida en y que es un dask array:
print(type(y))
<class 'dask.array.core.Array'>
y.visualize()
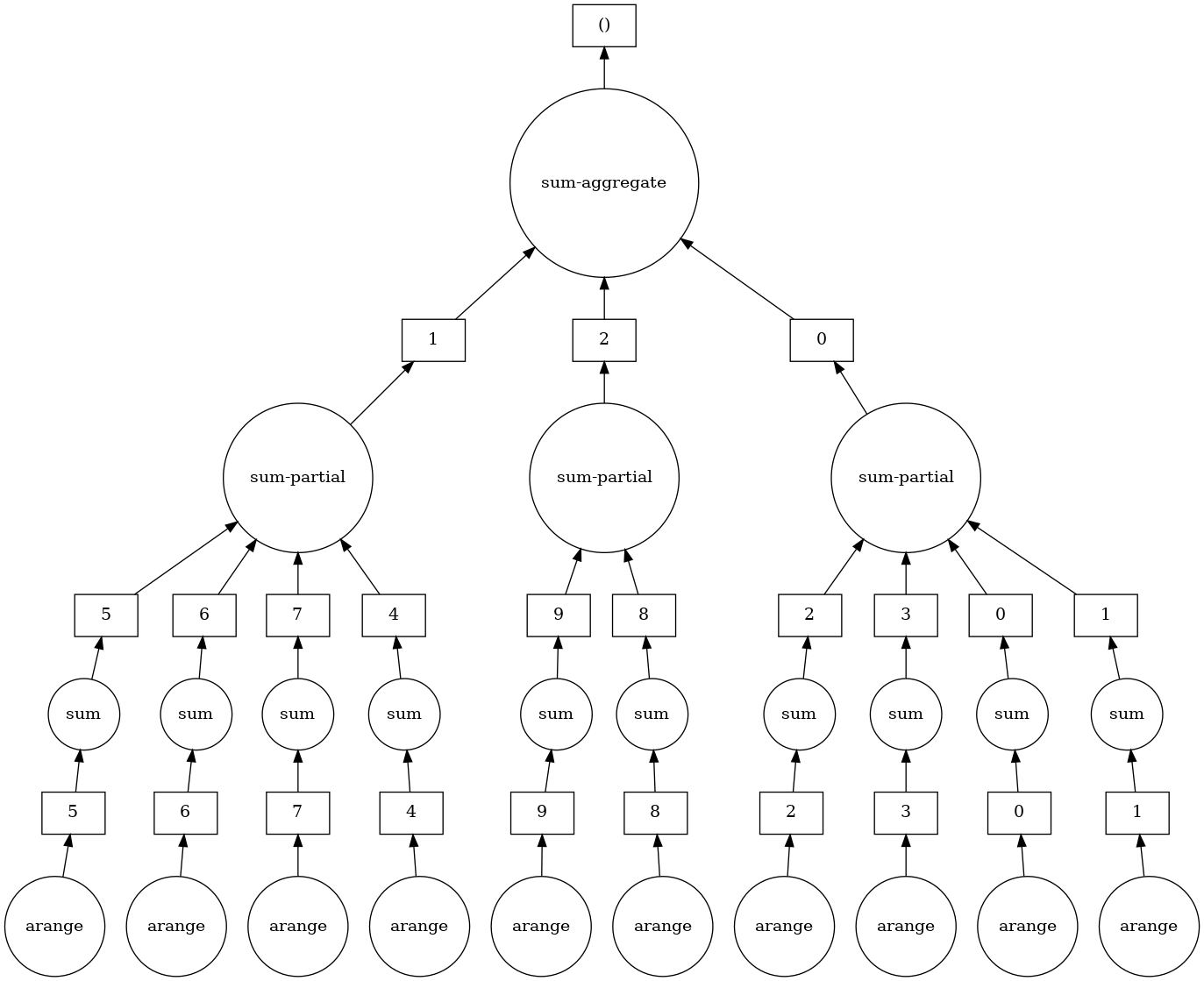
Ver la API de dask array en array api para más funcionalidad.
Comentario
Dask tiene una función compute más general que toma múltiples collections y regresa múltiples resultados. Esto combina grafos de cada collection de modo que resultados intermedios son compartidos.
y = (x+1).sum()
z = (x+1).mean()
print(da.compute(y, z))
(5050, 50.5)
y aquí el resultado intermedio x+1 sólo fue calculado una vez. Si hubiéramos hecho:
y.compute()
z.compute()
el resultado intermedio x+1 hubiera sido calculado dos veces. Ésta función compute trabaja sobre cualquier collection y se encuentra en dask.base:
from dask.base import compute
print(compute is da.compute)
True
print(compute(y,z))
(5050, 50.5)
También se puede especificar para un scheduler threaded o multiprocessing, el número de workers en compute:
print(y.compute(num_workers=2))
5050
Ver configuration para otras formas de especificar al scheduler. Ver también Delayed y Custom Workloads with Dask Delayed para otra opción diferente a los dask array’s, dask dataframe’s y dask bag’s para cómputo lazy.
Ejemplo regla compuesta del rectángulo con dask.array#
import numpy as np
f_np = lambda x: np.exp(-x**2)
def Rcf_dask_array(f,a,b,n, n_s_w, n_workers):
"""
Compute numerical approximation using rectangle or mid-point
method in an interval.
Nodes are generated via formula: x_i = a+(i+1/2)h_hat for
i=0,1,...,n-1 and h_hat=(b-a)/n
Args:
f (float): function expression of integrand.
a (float): left point of interval.
b (float): right point of interval.
n (int): number of subintervals.
n_s_w (int): number of subintervals per worker
n_workers (int): number of workers
Returns:
sum_res (float): numerical approximation to integral
of f in the interval a,b
"""
h_hat = (b-a)/n
aux_dask_array = da.linspace(a, b, n+1, chunks = n_s_w)
nodes = (aux_dask_array[:-1]+aux_dask_array[1:])/2
f_evaluated_in_nodes = f_np(nodes)
res_sum = f_evaluated_in_nodes.sum().compute(num_workers=n_workers)
return h_hat*res_sum
print(p, n_subintervals_per_subprocess)
64 156250
start_time=time.time()
res_rcf_dask_array = Rcf_dask_array(f_np,a,b,n,
n_subintervals_per_subprocess,
p)
end_time = time.time()
secs = end_time-start_time
print("Rcf_dask_array tomó",secs,"segundos" )
Rcf_dask_array tomó 0.1601552963256836 segundos
print(res_rcf_dask_array == approx(obj))
True
Dask distributed#
Es un paquete que extiende a concurrent.futures (
multiprocessinges similar aconcurrent.futures) y a Dask para clústers de tamaño mediano (alrededor de cientos de máquinas).Aunque el nombre distributed puede tomarse como que la ejecución sólo puede llevarse a cabo en clústers de máquinas, también puede utilizarse en nuestra máquina local.
Por razones como proveer acceso a una API asíncrona: Futures, acceso a un dashboard diagnóstico y manejar mejor la data locality con múltiples procesos más eficientemente que con
dask.multiprocessing.get, representa una alternativa fuerte a la generación de procesos locales víadask.multiprocessing: The distributed scheduler described a couple sections below is often a better choice today thandask.multiprocessing…
Tiene las siguientes características.
Low latency: Each task suffers about 1ms of overhead. A small computation and network roundtrip can complete in less than 10ms.
Peer-to-peer data sharing: Workers communicate with each other to share data. This removes central bottlenecks for data transfer.
Complex Scheduling: Supports complex workflows (not just map/filter/reduce) which are necessary for sophisticated algorithms used in nd-arrays, machine learning, image processing, and statistics.
Data Locality: Scheduling algorithms cleverly execute computations where data lives. This minimizes network traffic and improves efficiency.
Familiar APIs: Compatible with the concurrent.futures API in the Python standard library. Compatible with dask API for parallel algorithms.
Para ver más sobre el distributed scheduler ver dask-architecture.
¿concurrent.futures?#
Es un paquete para ejecución de tasks en paralelo.
En
concurrent.futuresse trabaja con objetos de tipo valorfuturelos cuales son resultados de cálculos que estarán disponibles en un futuro, de aquí el nombre de future y su cómputo no es lazy sino inmediato.La obtención del future es asíncrona por lo que permite cómputo concurrente.
Ejemplo#
import time
def inc(x):
time.sleep(1)
return x+1
Ejecución secuencial
%%time
inputs = [1,2,3,4,5,6,7,8,9,10]
results=[]
for x in inputs:
result=inc(x)
results.append(result)
CPU times: user 949 µs, sys: 1.02 ms, total: 1.97 ms
Wall time: 10 s
print(results)
[2, 3, 4, 5, 6, 7, 8, 9, 10, 11]
Ejecución con concurrent.futures
from concurrent.futures import ProcessPoolExecutor
import multiprocessing
executor = ProcessPoolExecutor(multiprocessing.cpu_count())
print(executor)
<concurrent.futures.process.ProcessPoolExecutor object at 0x7fd2dece6ca0>
%%time
future = executor.submit(inc,3)
CPU times: user 39.7 ms, sys: 189 ms, total: 229 ms
Wall time: 218 ms
Observación
Obsérvese que se obtiene el future inmediatamente. Ver submit
print(future)
<Future at 0x7fd2df547af0 state=running>
Observación
La ejecución del future se encuentra en algún subproceso del proceso principal. Obsérvese que tiene un estado running. Y después de un tiempo el estado ya es finished.
print(future)
<Future at 0x7fd2df547af0 state=running>
Para obtener el resultado hacemos:
print(future.result())
4
Entonces podemos hacer submit de varios cálculos de forma asíncrona con el objeto executor que devolverá futures:
%%time
inputs = [1,2,3,4,5,6,7,8,9,10]
futures=[]
for x in inputs:
future=executor.submit(inc,x)
futures.append(future)
results = [future.result() for future in futures]
CPU times: user 2.82 ms, sys: 3.09 ms, total: 5.92 ms
Wall time: 1.01 s
Comentario
El statement results = [future.result() for future in futures] bloquea la ejecución del proceso local hasta que todos los procesos finalizan la ejecución.
Observación
Obsérvese que se redujo el tiempo pues la ejecución fue en paralelo por los subprocesos que ejecutaron inc.
print(results)
[2, 3, 4, 5, 6, 7, 8, 9, 10, 11]
executor.shutdown()
Comentarios
También en
concurrent.futuresse tiene una función map para cómputo inmediato (no lazy) usando futures que soporta múltiples llamados a una función utilizando un iterable.Para una vista rápida a
concurrent.futuresver: Python: A quick introduction to the concurrent.futures module.
Client de dask.distributed#
El Client es el entry point para users de dask.distributed. Creamos un distributed scheduler al importar Client. Esta acción override cualquier configuración que se haya hecho del scheduler.
from dask.distributed import Client
client = Client()
Comentario
Al Client se le pueden pasar argumentos como número de workers, si se desean procesos o no, el límite de memoria a usar por cada proceso, entre otros.
print(client)
<Client: 'tcp://127.0.0.1:45025' processes=8 threads=64, memory=251.89 GiB>
client
Client
|
Cluster
|
Para ir al dashboard que se expone con bokeh ir a la url con puerto 8787.
Observación
Si se ejecuta con una imagen de Docker, revisar a qué puerto está mapeado el 8787.
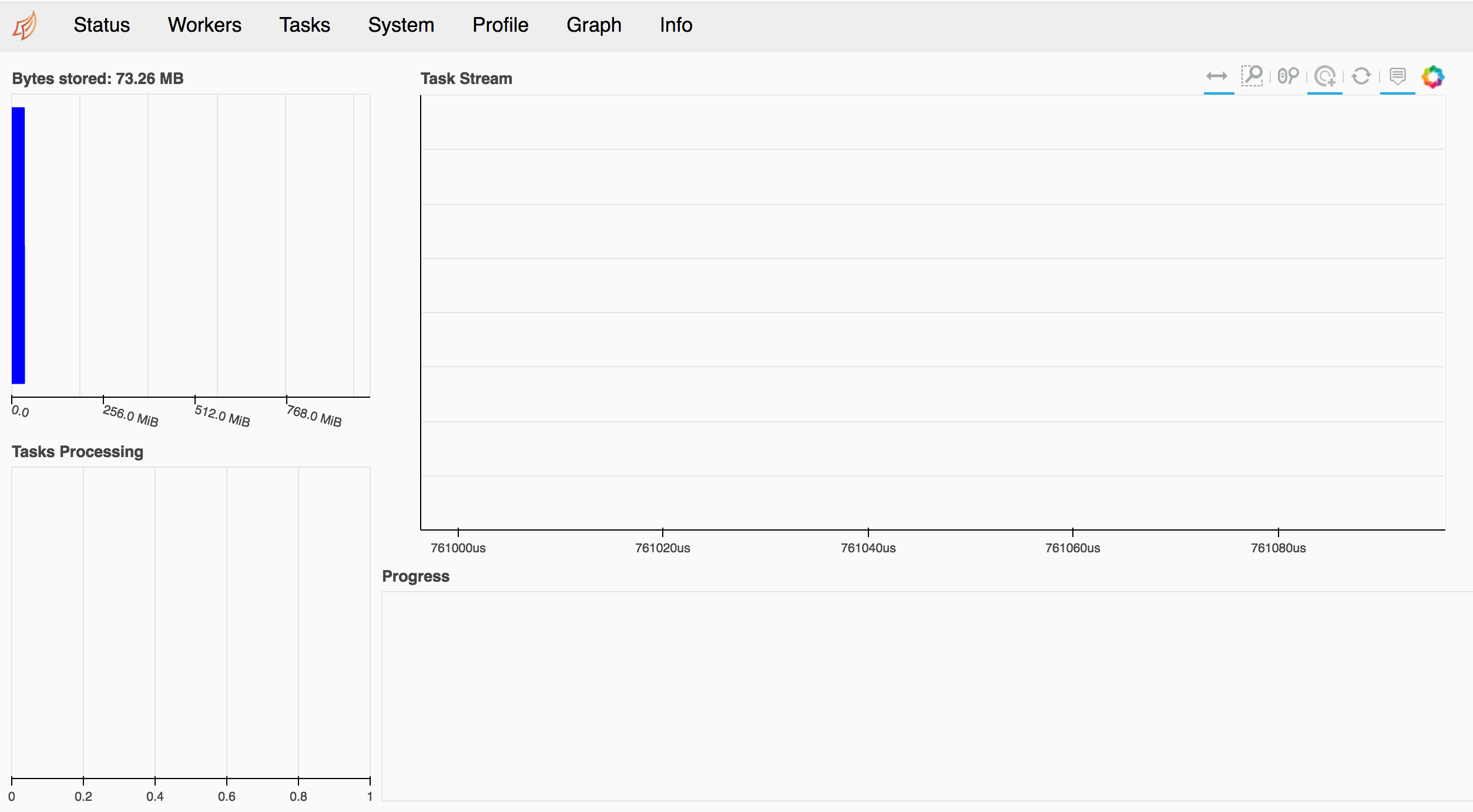
y en el apartado de workers:
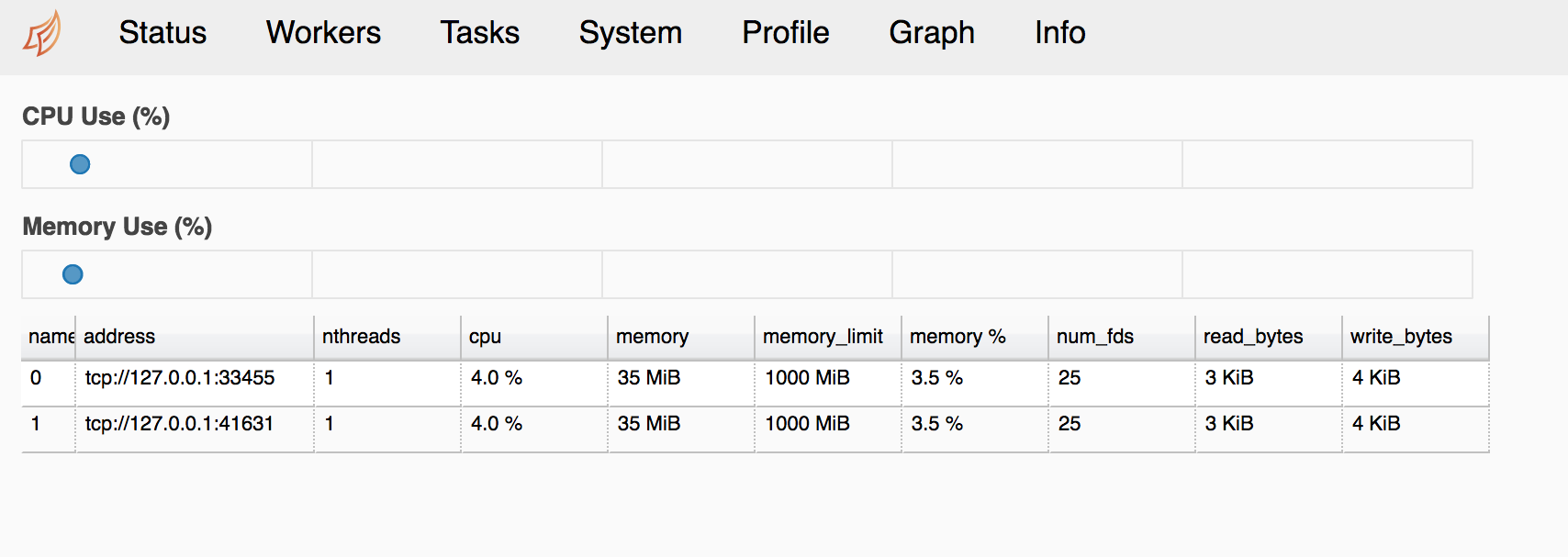
Ver Web Interface y Diagnosing Performance para más sobre la información de diagnóstico que se junta en el dashboard.
Comentarios
La línea anterior que contiene
Client, configura un scheduler en el proceso local y varios procesos workers o dask-workers que corren un thread de ejecución (pueden correr más por default). Si se desea que los procesos worker estén contenidos en el proceso local puede usarseClient(processes=False). Esta situación es preferible si se desea evitar comunicación inter worker y si nuestros cálculos están soltando el GIL (por ejemplo al usar NumPy odask.array).Algunas opciones que pueden utilizarse con la línea de
Clientson:Client(n_workers=4).Client(threads_per_worker=2).Client(memory_limit='10GB').
Ver LocalCluster para opciones a usar en Client y API para la API con Client.
Ejemplo regla compuesta del rectángulo con map y gather#
def create_nodes_in_subintervals(my_id,a,b,h_hat,n,n_s_sub):
begin = my_id*n_s_sub
end = begin+n_s_sub
h_hat = (b-a)/n
nodes = []
for i in range(begin,end):
nodes.append(a+(i+1/2.0)*h_hat)
return nodes
def evaluate_f_in_nodes_and_sum(nodes,f):
sum_res = 0
for node in nodes:
sum_res+=f(node)
return sum_res
%%time
futures_nodes = client.map(create_nodes_in_subintervals,range(p),
**{'a':a,
'b':b,
"h_hat":h_hat,
'n':n,
"n_s_sub":n_subintervals_per_subprocess}
)
futures_evaluated = client.map(evaluate_f_in_nodes_and_sum, futures_nodes,
**{'f':f}
)
results_gather = client.gather(futures_evaluated)
res_rcf_distributed = h_hat*sum(results_gather)
CPU times: user 110 ms, sys: 55.8 ms, total: 166 ms
Wall time: 1 s
print(res_rcf_distributed == approx(obj))
True
Comentarios
Con
maposubmitse han enviado las tasks al scheduler a realizar. Ver map, submit y concurrent.futures.Las ejecuciones
mapson muy rápidas e inmediatas pues se regresan futures.Los cálculos definidos por las funciones
create_nodes_in_subintervalsyevaluate_f_in_nodes_and_sumse ejecutan en cada dask-worker y a distintos tiempos.Obsérvese que se puede hacer
maposubmiten futures.Los resultados viven en los procesos dask-workers.
Con
gatherse juntan los resultados (futures) en el proceso local.Podemos usar
gatheroresultde los futures.
client.close()
Ejemplo regla compuesta del rectángulo con map y result#
client = Client(n_workers=multiprocessing.cpu_count(),
threads_per_worker=1
)
client
Client
|
Cluster
|
%%time
futures_nodes = client.map(create_nodes_in_subintervals,range(p),
**{'a':a,
'b':b,
"h_hat":h_hat,
'n':n,
"n_s_sub":n_subintervals_per_subprocess}
)
futures_evaluated = client.map(evaluate_f_in_nodes_and_sum, futures_nodes,
**{'f':f}
)
results = [future.result() for future in futures_evaluated]
res_rcf_distributed = h_hat*sum(results)
CPU times: user 428 ms, sys: 75.4 ms, total: 503 ms
Wall time: 630 ms
print(res_rcf_distributed == approx(obj))
True
Gráfica de tiempo de ejecución vs número de procesos#
err_np = []
n_cpus = []
def relative_error(aprox, obj):
return math.fabs(aprox-obj)/math.fabs(obj)
def Rcf_dask(my_id,f,a,b,h_hat,n,n_s_sub):
begin=my_id*n_s_sub
end=begin+n_s_sub
h_hat=(b-a)/n
sum_res = 0
for i in range(begin,end):
x = a+(i+1/2)*h_hat
sum_res += f(x)
return sum_res
def myfun(cl,p,n_s_sub):
futures_Rcf_dask = cl.map(Rcf_dask,range(p),
**{'f':f,
'a':a,
'b':b,
"h_hat":h_hat,
'n':n,
"n_s_sub":n_s_sub}
)
results_gather = cl.gather(futures_Rcf_dask)
res_rcf_dask = h_hat*sum(results_gather)
return relative_error(res_rcf_dask,obj)
client.close()
client
Client
|
Cluster
|
Primero revisamos errores relativos
for p in range(1,multiprocessing.cpu_count()+1):
if n%p==0:
n_subintervals_per_subprocess = int(n/p)
client = Client(n_workers=p,
threads_per_worker=1
)
err_np.append(myfun(client,p,n_subintervals_per_subprocess))
n_cpus.append(p)
client.close()
print(err_np)
[4.99495021497577e-14, 1.4419945561090765e-14, 2.4231454911936028e-14, 5.886905610507158e-14, 1.3825308630736507e-14, 1.7690448678039188e-14, 3.716480814714115e-15, 3.86514004730268e-15, 2.527206954005598e-15, 2.973184651771292e-15, 2.229888488828469e-15, 2.6758661865941626e-15, 1.486592325885646e-15]
print(n_cpus)
[1, 2, 4, 5, 8, 10, 16, 20, 25, 32, 40, 50, 64]
l=[]
n_cpus=[]
client.close()
client
Client
|
Cluster
|
for p in range(1,multiprocessing.cpu_count()+1):
if n%p==0:
n_subintervals_per_subprocess = int(n/p)
client = Client(n_workers=p,
threads_per_worker=1
)
start_time=time.time()
futures_Rcf_dask = client.map(Rcf_dask,range(p),
**{'f':f,
'a':a,
'b':b,
"h_hat":h_hat,
'n':n,
"n_s_sub":n_subintervals_per_subprocess}
)
res_gather = client.gather(futures_Rcf_dask)
end_time = time.time()
secs = end_time-start_time
l.append(secs)
n_cpus.append(p)
client.close()
print(l)
[3.701357364654541, 1.8432807922363281, 0.9091541767120361, 0.7361886501312256, 0.4691917896270752, 0.4330434799194336, 0.26079630851745605, 0.23067212104797363, 0.2382211685180664, 0.3052539825439453, 0.2685668468475342, 0.2464737892150879, 0.2692685127258301]
print(n_cpus)
[1, 2, 4, 5, 8, 10, 16, 20, 25, 32, 40, 50, 64]
import matplotlib.pyplot as plt
plt.plot(n_cpus,l,'o-')
plt.title('Gráfica num cpus vs tiempo')
plt.xlabel('num cpus')
plt.ylabel('tiempo')
plt.grid()
plt.show()
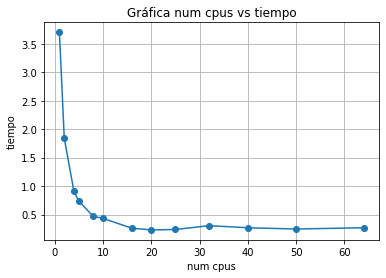
Ejercicio
Implementar la regla de Simpson compuesta con Multiprocessing y Dask en una máquina de AWS con las mismas características que la que se presenta en esta nota, medir tiempo de ejecución y realizar gráfica de tiempo vs num cpus.
Parallel#
Entre las herramientas más populares en R para cómputo en paralelo se encuentran:
Simple Network Of Workstations: snow. Ya es una herramienta incluida en el paquete Parallel). Ver liga para más información.
multicore. Funciona en la familia Unix pero no en Windows y ya es una herramienta incluida en el paquete Parallel.
Rmpi. Paralelización en SMC y en SMD.
Comentarios
Las primeras dos son parte del paquete parallel el cual está incluido en la instalación de R desde la versión
2.14.0.Los cuatro paquetes de arriba emplean un paradigma de programación en paralelo del tipo scatter/gather: se tienen múltiples instancias de R corriendo al mismo tiempo en un clúster de máquinas o en una máquina multicore. Una de las instancias se le denomina manager y las restantes workers. El cómputo en paralelo procede como sigue:
scatter: manager descompone el cómputo a realizar en chunks y envía (scatters) los chunks a workers.
chunk computation: workers hacen el cómputo en cada chunk y envían de regreso los resultados a manager.
gather: manager recibe (gathers) los resultados y los combina para resolver el problema.
Ejemplo#
library(parallel)
En Parallel tenemos la función detectCores que como su nombre lo indica obtiene el número de CPU’s en nuestra máquina.
p <- detectCores()
p
Usamos makeCluster para iniciar el snow cluster:
cl <- makeCluster(p)
La línea anterior crea p workers y cada worker es un proceso de R (al igual que el proceso manager) que corren en la misma máquina.
Comentario
El nombre de clúster en snow hace referencia al conjunto de workers y no a máquinas físicas. El objeto cl contiene información de workers y es de clase cluster.
print(class(cl))
[1] "SOCKcluster" "cluster"
La comunicación entre manager y workers por default en makeCluster es vía network sockets y es posible crear un clúster de máquinas físicas conectadas vía una network.
print(cl)
socket cluster with 64 nodes on host ‘localhost’
clusterApply#
En Parallel se tiene la función clusterApply para ejecución en paralelo de una función aplicada a cada elemento de una lista: manager envía lista[1] a cl[1], lista[2] a cl[2],…,lista[p] a cl[p].
Además, si el número de elementos de la lista es mayor que el número de workers entonces clusterApply realiza una asignación tipo round robin: por ejemplo, si la lista tiene \(4\) elementos y se tienen \(2\) workers entonces clusterApply le envía a worker1 el elemento de la primera posición de la lista, a worker2 el segundo elemento. Después que worker1 finaliza el procesamiento, clusterApply le envía la tercera posición y al terminar worker2 del procesamiento, clusterApply le envía la cuarta posición (clusterApply aprovecha la característica que tiene R de reciclar operaciones).
print(clusterApply(cl, 1:p,function(dummy)print("Hello world!")))
[[1]]
[1] "Hello world!"
[[2]]
[1] "Hello world!"
[[3]]
[1] "Hello world!"
[[4]]
[1] "Hello world!"
[[5]]
[1] "Hello world!"
[[6]]
[1] "Hello world!"
[[7]]
[1] "Hello world!"
[[8]]
[1] "Hello world!"
[[9]]
[1] "Hello world!"
[[10]]
[1] "Hello world!"
[[11]]
[1] "Hello world!"
[[12]]
[1] "Hello world!"
[[13]]
[1] "Hello world!"
[[14]]
[1] "Hello world!"
[[15]]
[1] "Hello world!"
[[16]]
[1] "Hello world!"
[[17]]
[1] "Hello world!"
[[18]]
[1] "Hello world!"
[[19]]
[1] "Hello world!"
[[20]]
[1] "Hello world!"
[[21]]
[1] "Hello world!"
[[22]]
[1] "Hello world!"
[[23]]
[1] "Hello world!"
[[24]]
[1] "Hello world!"
[[25]]
[1] "Hello world!"
[[26]]
[1] "Hello world!"
[[27]]
[1] "Hello world!"
[[28]]
[1] "Hello world!"
[[29]]
[1] "Hello world!"
[[30]]
[1] "Hello world!"
[[31]]
[1] "Hello world!"
[[32]]
[1] "Hello world!"
[[33]]
[1] "Hello world!"
[[34]]
[1] "Hello world!"
[[35]]
[1] "Hello world!"
[[36]]
[1] "Hello world!"
[[37]]
[1] "Hello world!"
[[38]]
[1] "Hello world!"
[[39]]
[1] "Hello world!"
[[40]]
[1] "Hello world!"
[[41]]
[1] "Hello world!"
[[42]]
[1] "Hello world!"
[[43]]
[1] "Hello world!"
[[44]]
[1] "Hello world!"
[[45]]
[1] "Hello world!"
[[46]]
[1] "Hello world!"
[[47]]
[1] "Hello world!"
[[48]]
[1] "Hello world!"
[[49]]
[1] "Hello world!"
[[50]]
[1] "Hello world!"
[[51]]
[1] "Hello world!"
[[52]]
[1] "Hello world!"
[[53]]
[1] "Hello world!"
[[54]]
[1] "Hello world!"
[[55]]
[1] "Hello world!"
[[56]]
[1] "Hello world!"
[[57]]
[1] "Hello world!"
[[58]]
[1] "Hello world!"
[[59]]
[1] "Hello world!"
[[60]]
[1] "Hello world!"
[[61]]
[1] "Hello world!"
[[62]]
[1] "Hello world!"
[[63]]
[1] "Hello world!"
[[64]]
[1] "Hello world!"
print(clusterApply(cl, 1:5,function(dummy)print("Hello world!")))
[[1]]
[1] "Hello world!"
[[2]]
[1] "Hello world!"
[[3]]
[1] "Hello world!"
[[4]]
[1] "Hello world!"
[[5]]
[1] "Hello world!"
Observación
Obsérvese que el resultado de clusterApply es una lista.
Ejemplo de pasar argumentos vía clusterExport#
clusterExport nos permite enviar valores de variables definidas en el workspace global de manager hacia los workspaces de cada worker bajo los mismos nombres de variables:
s <- "Hola mundo!"
clusterExport(cl,'s')
print(clusterApply(cl, 1:p,function(dummy)print(s)))
[[1]]
[1] "Hola mundo!"
[[2]]
[1] "Hola mundo!"
[[3]]
[1] "Hola mundo!"
[[4]]
[1] "Hola mundo!"
[[5]]
[1] "Hola mundo!"
[[6]]
[1] "Hola mundo!"
[[7]]
[1] "Hola mundo!"
[[8]]
[1] "Hola mundo!"
[[9]]
[1] "Hola mundo!"
[[10]]
[1] "Hola mundo!"
[[11]]
[1] "Hola mundo!"
[[12]]
[1] "Hola mundo!"
[[13]]
[1] "Hola mundo!"
[[14]]
[1] "Hola mundo!"
[[15]]
[1] "Hola mundo!"
[[16]]
[1] "Hola mundo!"
[[17]]
[1] "Hola mundo!"
[[18]]
[1] "Hola mundo!"
[[19]]
[1] "Hola mundo!"
[[20]]
[1] "Hola mundo!"
[[21]]
[1] "Hola mundo!"
[[22]]
[1] "Hola mundo!"
[[23]]
[1] "Hola mundo!"
[[24]]
[1] "Hola mundo!"
[[25]]
[1] "Hola mundo!"
[[26]]
[1] "Hola mundo!"
[[27]]
[1] "Hola mundo!"
[[28]]
[1] "Hola mundo!"
[[29]]
[1] "Hola mundo!"
[[30]]
[1] "Hola mundo!"
[[31]]
[1] "Hola mundo!"
[[32]]
[1] "Hola mundo!"
[[33]]
[1] "Hola mundo!"
[[34]]
[1] "Hola mundo!"
[[35]]
[1] "Hola mundo!"
[[36]]
[1] "Hola mundo!"
[[37]]
[1] "Hola mundo!"
[[38]]
[1] "Hola mundo!"
[[39]]
[1] "Hola mundo!"
[[40]]
[1] "Hola mundo!"
[[41]]
[1] "Hola mundo!"
[[42]]
[1] "Hola mundo!"
[[43]]
[1] "Hola mundo!"
[[44]]
[1] "Hola mundo!"
[[45]]
[1] "Hola mundo!"
[[46]]
[1] "Hola mundo!"
[[47]]
[1] "Hola mundo!"
[[48]]
[1] "Hola mundo!"
[[49]]
[1] "Hola mundo!"
[[50]]
[1] "Hola mundo!"
[[51]]
[1] "Hola mundo!"
[[52]]
[1] "Hola mundo!"
[[53]]
[1] "Hola mundo!"
[[54]]
[1] "Hola mundo!"
[[55]]
[1] "Hola mundo!"
[[56]]
[1] "Hola mundo!"
[[57]]
[1] "Hola mundo!"
[[58]]
[1] "Hola mundo!"
[[59]]
[1] "Hola mundo!"
[[60]]
[1] "Hola mundo!"
[[61]]
[1] "Hola mundo!"
[[62]]
[1] "Hola mundo!"
[[63]]
[1] "Hola mundo!"
[[64]]
[1] "Hola mundo!"
¿Qué pasa si mi clusterExport está dentro de una función?
myfun <- function(){
s_local<-"Hola mundo! usando variable local a myfun"
clusterExport(cl,"s_local")
print(clusterApply(cl, 1:p,function(dummy)print(s_local)))
}
print(myfun())
Error in get(name, envir = envir): object 's_local' not found
Traceback:
1. print(myfun())
2. myfun()
3. clusterExport(cl, "s_local") # at line 3 of file <text>
4. clusterCall(cl, gets, name, get(name, envir = envir))
5. sendCall(cl[[i]], fun, list(...))
6. postNode(con, "EXEC", list(fun = fun, args = args, return = return,
. tag = tag))
7. sendData(con, list(type = type, data = value, tag = tag))
8. sendData.SOCKnode(con, list(type = type, data = value, tag = tag))
9. serialize(data, node$con)
10. get(name, envir = envir)
Lo anterior lo podemos resolver con superassignment. Ver What does the “<<-” operator mean? y how-do-you-use-scoping-assignment-in-r.
myfun2 <- function(){
s_global <<- "Hola mundo! usando superassignment en mifun2" #<<- superassignment
clusterExport(cl,"s_global")
clusterApply(cl, 1:p,function(dummy)print(s_global))
}
print(myfun2())
[[1]]
[1] "Hola mundo! usando superassignment en mifun2"
[[2]]
[1] "Hola mundo! usando superassignment en mifun2"
[[3]]
[1] "Hola mundo! usando superassignment en mifun2"
[[4]]
[1] "Hola mundo! usando superassignment en mifun2"
[[5]]
[1] "Hola mundo! usando superassignment en mifun2"
[[6]]
[1] "Hola mundo! usando superassignment en mifun2"
[[7]]
[1] "Hola mundo! usando superassignment en mifun2"
[[8]]
[1] "Hola mundo! usando superassignment en mifun2"
[[9]]
[1] "Hola mundo! usando superassignment en mifun2"
[[10]]
[1] "Hola mundo! usando superassignment en mifun2"
[[11]]
[1] "Hola mundo! usando superassignment en mifun2"
[[12]]
[1] "Hola mundo! usando superassignment en mifun2"
[[13]]
[1] "Hola mundo! usando superassignment en mifun2"
[[14]]
[1] "Hola mundo! usando superassignment en mifun2"
[[15]]
[1] "Hola mundo! usando superassignment en mifun2"
[[16]]
[1] "Hola mundo! usando superassignment en mifun2"
[[17]]
[1] "Hola mundo! usando superassignment en mifun2"
[[18]]
[1] "Hola mundo! usando superassignment en mifun2"
[[19]]
[1] "Hola mundo! usando superassignment en mifun2"
[[20]]
[1] "Hola mundo! usando superassignment en mifun2"
[[21]]
[1] "Hola mundo! usando superassignment en mifun2"
[[22]]
[1] "Hola mundo! usando superassignment en mifun2"
[[23]]
[1] "Hola mundo! usando superassignment en mifun2"
[[24]]
[1] "Hola mundo! usando superassignment en mifun2"
[[25]]
[1] "Hola mundo! usando superassignment en mifun2"
[[26]]
[1] "Hola mundo! usando superassignment en mifun2"
[[27]]
[1] "Hola mundo! usando superassignment en mifun2"
[[28]]
[1] "Hola mundo! usando superassignment en mifun2"
[[29]]
[1] "Hola mundo! usando superassignment en mifun2"
[[30]]
[1] "Hola mundo! usando superassignment en mifun2"
[[31]]
[1] "Hola mundo! usando superassignment en mifun2"
[[32]]
[1] "Hola mundo! usando superassignment en mifun2"
[[33]]
[1] "Hola mundo! usando superassignment en mifun2"
[[34]]
[1] "Hola mundo! usando superassignment en mifun2"
[[35]]
[1] "Hola mundo! usando superassignment en mifun2"
[[36]]
[1] "Hola mundo! usando superassignment en mifun2"
[[37]]
[1] "Hola mundo! usando superassignment en mifun2"
[[38]]
[1] "Hola mundo! usando superassignment en mifun2"
[[39]]
[1] "Hola mundo! usando superassignment en mifun2"
[[40]]
[1] "Hola mundo! usando superassignment en mifun2"
[[41]]
[1] "Hola mundo! usando superassignment en mifun2"
[[42]]
[1] "Hola mundo! usando superassignment en mifun2"
[[43]]
[1] "Hola mundo! usando superassignment en mifun2"
[[44]]
[1] "Hola mundo! usando superassignment en mifun2"
[[45]]
[1] "Hola mundo! usando superassignment en mifun2"
[[46]]
[1] "Hola mundo! usando superassignment en mifun2"
[[47]]
[1] "Hola mundo! usando superassignment en mifun2"
[[48]]
[1] "Hola mundo! usando superassignment en mifun2"
[[49]]
[1] "Hola mundo! usando superassignment en mifun2"
[[50]]
[1] "Hola mundo! usando superassignment en mifun2"
[[51]]
[1] "Hola mundo! usando superassignment en mifun2"
[[52]]
[1] "Hola mundo! usando superassignment en mifun2"
[[53]]
[1] "Hola mundo! usando superassignment en mifun2"
[[54]]
[1] "Hola mundo! usando superassignment en mifun2"
[[55]]
[1] "Hola mundo! usando superassignment en mifun2"
[[56]]
[1] "Hola mundo! usando superassignment en mifun2"
[[57]]
[1] "Hola mundo! usando superassignment en mifun2"
[[58]]
[1] "Hola mundo! usando superassignment en mifun2"
[[59]]
[1] "Hola mundo! usando superassignment en mifun2"
[[60]]
[1] "Hola mundo! usando superassignment en mifun2"
[[61]]
[1] "Hola mundo! usando superassignment en mifun2"
[[62]]
[1] "Hola mundo! usando superassignment en mifun2"
[[63]]
[1] "Hola mundo! usando superassignment en mifun2"
[[64]]
[1] "Hola mundo! usando superassignment en mifun2"
O bien con environment():
myfun3 <- function(s_global2){
clusterExport(cl,"s_global2")
clusterApply(cl, 1:p,function(dummy)print(s_global2))
}
print(myfun3("Hola mundo! usando variable local a mifun3"))
Error in get(name, envir = envir): object 's_global2' not found
Traceback:
1. print(myfun3("Hola mundo! usando variable local a mifun3"))
2. myfun3("Hola mundo! usando variable local a mifun3")
3. clusterExport(cl, "s_global2") # at line 2 of file <text>
4. clusterCall(cl, gets, name, get(name, envir = envir))
5. sendCall(cl[[i]], fun, list(...))
6. postNode(con, "EXEC", list(fun = fun, args = args, return = return,
. tag = tag))
7. sendData(con, list(type = type, data = value, tag = tag))
8. sendData.SOCKnode(con, list(type = type, data = value, tag = tag))
9. serialize(data, node$con)
10. get(name, envir = envir)
myfun4<-function(s_global2){
clusterExport(cl,"s_global2", envir=environment())
clusterApply(cl, 1:p,function(dummy)print(s_global2))
}
print(myfun4("Hola mundo! usando environment()"))
[[1]]
[1] "Hola mundo! usando environment()"
[[2]]
[1] "Hola mundo! usando environment()"
[[3]]
[1] "Hola mundo! usando environment()"
[[4]]
[1] "Hola mundo! usando environment()"
[[5]]
[1] "Hola mundo! usando environment()"
[[6]]
[1] "Hola mundo! usando environment()"
[[7]]
[1] "Hola mundo! usando environment()"
[[8]]
[1] "Hola mundo! usando environment()"
[[9]]
[1] "Hola mundo! usando environment()"
[[10]]
[1] "Hola mundo! usando environment()"
[[11]]
[1] "Hola mundo! usando environment()"
[[12]]
[1] "Hola mundo! usando environment()"
[[13]]
[1] "Hola mundo! usando environment()"
[[14]]
[1] "Hola mundo! usando environment()"
[[15]]
[1] "Hola mundo! usando environment()"
[[16]]
[1] "Hola mundo! usando environment()"
[[17]]
[1] "Hola mundo! usando environment()"
[[18]]
[1] "Hola mundo! usando environment()"
[[19]]
[1] "Hola mundo! usando environment()"
[[20]]
[1] "Hola mundo! usando environment()"
[[21]]
[1] "Hola mundo! usando environment()"
[[22]]
[1] "Hola mundo! usando environment()"
[[23]]
[1] "Hola mundo! usando environment()"
[[24]]
[1] "Hola mundo! usando environment()"
[[25]]
[1] "Hola mundo! usando environment()"
[[26]]
[1] "Hola mundo! usando environment()"
[[27]]
[1] "Hola mundo! usando environment()"
[[28]]
[1] "Hola mundo! usando environment()"
[[29]]
[1] "Hola mundo! usando environment()"
[[30]]
[1] "Hola mundo! usando environment()"
[[31]]
[1] "Hola mundo! usando environment()"
[[32]]
[1] "Hola mundo! usando environment()"
[[33]]
[1] "Hola mundo! usando environment()"
[[34]]
[1] "Hola mundo! usando environment()"
[[35]]
[1] "Hola mundo! usando environment()"
[[36]]
[1] "Hola mundo! usando environment()"
[[37]]
[1] "Hola mundo! usando environment()"
[[38]]
[1] "Hola mundo! usando environment()"
[[39]]
[1] "Hola mundo! usando environment()"
[[40]]
[1] "Hola mundo! usando environment()"
[[41]]
[1] "Hola mundo! usando environment()"
[[42]]
[1] "Hola mundo! usando environment()"
[[43]]
[1] "Hola mundo! usando environment()"
[[44]]
[1] "Hola mundo! usando environment()"
[[45]]
[1] "Hola mundo! usando environment()"
[[46]]
[1] "Hola mundo! usando environment()"
[[47]]
[1] "Hola mundo! usando environment()"
[[48]]
[1] "Hola mundo! usando environment()"
[[49]]
[1] "Hola mundo! usando environment()"
[[50]]
[1] "Hola mundo! usando environment()"
[[51]]
[1] "Hola mundo! usando environment()"
[[52]]
[1] "Hola mundo! usando environment()"
[[53]]
[1] "Hola mundo! usando environment()"
[[54]]
[1] "Hola mundo! usando environment()"
[[55]]
[1] "Hola mundo! usando environment()"
[[56]]
[1] "Hola mundo! usando environment()"
[[57]]
[1] "Hola mundo! usando environment()"
[[58]]
[1] "Hola mundo! usando environment()"
[[59]]
[1] "Hola mundo! usando environment()"
[[60]]
[1] "Hola mundo! usando environment()"
[[61]]
[1] "Hola mundo! usando environment()"
[[62]]
[1] "Hola mundo! usando environment()"
[[63]]
[1] "Hola mundo! usando environment()"
[[64]]
[1] "Hola mundo! usando environment()"
Ejemplo regla compuesta del rectángulo con clusterApply#
library(microbenchmark)
library(tictoc)
f <- function(x)exp(-x**2)
a <- 0
b <- 1
n <- 10**7
h_hat <- (b-a)/n
n_subintervals_per_subprocess <- as.integer(n/p)
Comentario
n_subintervals_per_subprocess es el número de subintervalos por core. Se asume que n es divisible por p. Si no se cumple esto, se puede definir n_subintervals_per_subprocess <- int(n/p) habiendo definido n primero y redefinir n como: n <- p*n_subintervals_per_subprocess
relative_error <- function(aprox,obj)abs(aprox-obj)/abs(obj)
sprintf("número de subintervalos: %d",n)
sprintf("número de subintervalos por proceso: %d",n_subintervals_per_subprocess)
Rcf_parallel_cl_apply <- function(my_id){
begin <- my_id*n_subintervals_per_subprocess
end <- begin+n_subintervals_per_subprocess
sum_res <- 0
for(j in begin:(end-1)){
x <- a+(j+1/2)*h_hat
sum_res <- sum_res+f(x)
}
sum_res
}
clusterExport(cl,c("n_subintervals_per_subprocess",'a','f',"h_hat"))
Utilizamos la función Reduce y la función sum a la lista que resulta de clusterApply:
tic("regla Rcf_parallel_cl_apply")
result_cl_apply <- clusterApply(cl,0:(p-1),Rcf_parallel_cl_apply)
res_rcf_cl_apply <- h_hat*Reduce(sum,result_cl_apply)
toc()
regla Rcf_parallel_cl_apply: 0.562 sec elapsed
obj <- integrate(Vectorize(f),a,b)
print(relative_error(res_rcf_cl_apply,obj$value))
[1] 1.486592e-15
La encapsulamos en una función para pasarla a microbenchmark:
clapply <- function(cl,p){
result_cl_apply <- clusterApply(cl,0:(p-1),Rcf_parallel_cl_apply)
res_rcf_cl_apply <- h_hat*Reduce(sum,result_cl_apply)
}
mbk<-microbenchmark(
clapply(cl,p),
times=10
)
print(mbk)
Unit: milliseconds
expr min lq mean median uq max neval
clapply(cl, p) 274.0143 299.3502 335.1274 322.0482 361.4857 457.578 10
Ejemplo regla compuesta del rectángulo con clusterSplit#
La función clusterSplit divide una secuencia en chunks de subsecuencias consecutivas. El número de chunks que regresa es igual al número de workers. clusterSplit intenta que los chunks sean similares en longitud y la división se realiza en manager.
En la siguiente implementación se recibe un chunk de nodos.
Rcf_parallel_cl_split<-function(chunk){
sum_res <- 0
for(x in chunk){
sum_res <- sum_res+f(x)
}
sum_res
}
tic()
#chunks for nodes using clusterSplit:
chunks <- clusterSplit(cl,vapply(0:(n-1),function(j)a+(j+1/2.0)*h_hat,numeric(1)))
#call clusterApply
result_cl_apply <- clusterApply(cl,chunks,Rcf_parallel_cl_split)
res_rcf_cl_split <- h_hat*Reduce(sum,result_cl_apply)
toc()
9.949 sec elapsed
print(relative_error(res_rcf_cl_split,obj$value))
[1] 2.824525e-15
En la siguiente implementación, se divide en chunks la secuencia: 0,1,...,n-1:
Rcf_parallel_cl_split_2 <- function(chunk){
n_s_sub <- length(chunk)
begin <- chunk[1]
end <- begin+n_s_sub
sum_res <- 0
for(j in begin:(end-1)){
x <- a+(j+1/2)*h_hat
sum_res <- sum_res+f(x)
}
sum_res
}
tic()
#se crean los chunks de la secuencia: 0,1,...,n-1
chunks <- clusterSplit(cl,0:(n-1))
result_cl_apply <- clusterApply(cl,chunks,Rcf_parallel_cl_split_2)
res_rcf_cl_split_2 <- h_hat*Reduce(sum,result_cl_apply)
toc()
1.538 sec elapsed
print(relative_error(res_rcf_cl_split_2,obj$value))
[1] 2.824525e-15
Medimos tiempos con Microbenchmark.
clapply2 <- function(cl){
chunks <- clusterSplit(cl,vapply(0:(n-1),function(j)a+(j+1/2.0)*h_hat,numeric(1)))
result_cl_apply <- clusterApply(cl,chunks,Rcf_parallel_cl_split)
res_rcf_cl_split <- h_hat*Reduce(sum,result_cl_apply)
}
clapply3 <- function(cl){
chunks <- clusterSplit(cl,0:(n-1))
result_cl_apply <- clusterApply(cl,chunks,Rcf_parallel_cl_split_2)
res_rcf_cl_split_2 <- h_hat*Reduce(sum,result_cl_apply)
}
mbk<-microbenchmark(
clapply(cl,p),
clapply2(cl),
clapply3(cl),
times=10
)
print(mbk)
Unit: milliseconds
expr min lq mean median uq max
clapply(cl, p) 279.1311 282.8535 305.9289 294.0048 325.548 371.0334
clapply2(cl) 9692.8472 9737.9523 10064.3089 9856.9876 9932.777 12093.3270
clapply3(cl) 1341.8627 1463.4982 1484.6908 1478.0374 1539.323 1618.6653
neval
10
10
10
Gráfica de tiempo de ejecución vs número de procesos#
library(ggplot2)
Una buena práctica es detener el clúster:
It is good practice to shut down the workers by calling stopCluster: however the workers will terminate themselves once the socket on which they are listening for commands becomes unavailable, which it should if the master R session is completed (or its process dies).
stopCluster(cl)
En lo siguiente se utiliza Rcf_parallel_cl_apply pues tuvo el menor tiempo de ejecución. Usaremos ésta implementación para realizar la gráfica variando el número de procesos de \(1\) hasta detectCores().
myfun <- function(cl,p){
result_apply <- clusterApply(cl,0:(p-1),Rcf_parallel_cl_apply)
res_rcf_cl_apply <- h_hat*Reduce(sum,result_apply)
}
dim <- sum(n%%(1:detectCores())==0)
err_vec <- vector("double", dim)
n_cpus <- vector("integer",dim)
i<-1
for(p in 1:detectCores()){
if(n%%p==0){
n_subintervals_per_subprocess <- n/p
cl <- makeCluster(p)
clusterExport(cl,c('a','f',"h_hat","n_subintervals_per_subprocess"))
res_my_fun <- myfun(cl,p)
err_vec[i] <- relative_error(res_my_fun, obj$value)
n_cpus[i] <- p
stopCluster(cl)
i <- i+1
}
}
print(err_vec)
[1] 4.994950e-14 1.441995e-14 2.423145e-14 5.886906e-14 1.382531e-14
[6] 1.769045e-14 3.716481e-15 3.865140e-15 2.527207e-15 2.973185e-15
[11] 2.229888e-15 2.675866e-15 1.486592e-15
print(n_cpus)
[1] 1 2 4 5 8 10 16 20 25 32 40 50 64
vector_with_time_medians <- vector("double", dim)
n_cpus <- vector("integer",dim)
i<-1
for(p in 1:detectCores()){
if(n%%p==0){
n_subintervals_per_subprocess <- n/p
cl <- makeCluster(p)
clusterExport(cl,c('a','f',"h_hat","n_subintervals_per_subprocess"))
df <- print(microbenchmark(myfun(cl,p),times=10,unit="s"))
stopCluster(cl)
vector_with_time_medians[i] <- df$median
n_cpus[i] <- p
i <- i+1
}
}
Unit: seconds
expr min lq mean median uq max neval
myfun(cl, p) 6.880437 6.912525 6.962728 6.951347 6.969166 7.092888 10
Unit: seconds
expr min lq mean median uq max neval
myfun(cl, p) 3.521054 3.528644 3.550711 3.537149 3.561584 3.63593 10
Unit: seconds
expr min lq mean median uq max neval
myfun(cl, p) 1.810182 1.84642 1.868893 1.855109 1.877132 1.992167 10
Unit: seconds
expr min lq mean median uq max neval
myfun(cl, p) 1.497631 1.503919 1.539663 1.513827 1.561826 1.68751 10
Unit: seconds
expr min lq mean median uq max neval
myfun(cl, p) 0.9574237 0.9681314 0.9938 0.9926238 1.002309 1.084198 10
Unit: seconds
expr min lq mean median uq max neval
myfun(cl, p) 0.7567488 0.7752932 0.8005973 0.7915049 0.8226106 0.8705034 10
Unit: seconds
expr min lq mean median uq max neval
myfun(cl, p) 0.4981435 0.5000754 0.5255358 0.5042411 0.5605582 0.6147569 10
Unit: seconds
expr min lq mean median uq max neval
myfun(cl, p) 0.4741282 0.6334634 0.6247839 0.644421 0.679061 0.7279318 10
Unit: seconds
expr min lq mean median uq max neval
myfun(cl, p) 0.4189323 0.5358796 0.5622284 0.5821805 0.6220992 0.6457455 10
Unit: seconds
expr min lq mean median uq max neval
myfun(cl, p) 0.5009026 0.5067084 0.526749 0.5106894 0.5534964 0.5992958 10
Unit: seconds
expr min lq mean median uq max neval
myfun(cl, p) 0.3703077 0.4051899 0.4655505 0.4660017 0.5055815 0.5712992 10
Unit: seconds
expr min lq mean median uq max neval
myfun(cl, p) 0.3289509 0.333303 0.3752226 0.3503937 0.3883032 0.5635342 10
Unit: seconds
expr min lq mean median uq max neval
myfun(cl, p) 0.2982448 0.3310383 0.3636902 0.3519018 0.3622468 0.5368318 10
print(vector_with_time_medians)
[1] 6.9513466 3.5371493 1.8551087 1.5138267 0.9926238 0.7915049 0.5042411
[8] 0.6444210 0.5821805 0.5106894 0.4660017 0.3503937 0.3519018
print(n_cpus)
[1] 1 2 4 5 8 10 16 20 25 32 40 50 64
gg<-ggplot()
gg+
geom_line(aes(x=n_cpus,y=vector_with_time_medians))
Ejercicio
Implementar la regla de Simpson compuesta con Parallel en una máquina de AWS con las mismas características que la que se presenta en esta nota, medir tiempo de ejecución y realizar gráfica de tiempo vs num cpus.
Referencias de interés#
matthew-rocklin-blog. Ver Towards-OutOfCore-ND-Arrays-Slicing-and-Stacking. Matthew Rocklin: Dask: out of core arrays with task scheduling
En dask se hace referencia al uso de funciones pure. Ver: Pure Functions by Default y Function Purity para ejemplos de funciones pure.
En Dask JupyterLab Extension se muestra cómo instalar la extensión en jupyterlab para Dask.
Paquetes de R que ayudan al cómputo paralelo y concurrente: future, Launching tasks with future y furrr
Ejercicios
1.Resuelve los ejercicios y preguntas de la nota.
Preguntas de comprehensión:
1)Menciona diferencias que surgen en un programa que se ejecuta en un sistema de memoria compartida contra los que se ejecutan en un sistema de memoria distribuida.
2)¿A qué se le llama no determinismo y da un ejemplo en el que esto surge en un sistema de memoria compartida?
3)¿Qué es una critical section? ¿qué es una race condition? ¿cómo se puede lidiar con las critical sections?
4)¿Cuál es la terminología para nombrar a las variables que pueden ser accesadas por todos los threads y para las variables que sólo pueden ser accesadas por un thread?
Referencias:
P. Pacheco, An Introduction to Parallel Programming, Morgan Kaufmann, 2011.
M. Gorelick, I. Ozsvald, High Performance Python, O’Reilly Media, 2014.
N. Matloff, Parallel Computing for Data Science. With Examples in R, C++ and CUDA, 2014.
B. W. Kernighan, D. M. Ritchie, The C Programming Language, Prentice Hall Software Series, 1988

